Autora: Melissa Markoski (@melmarkoski)
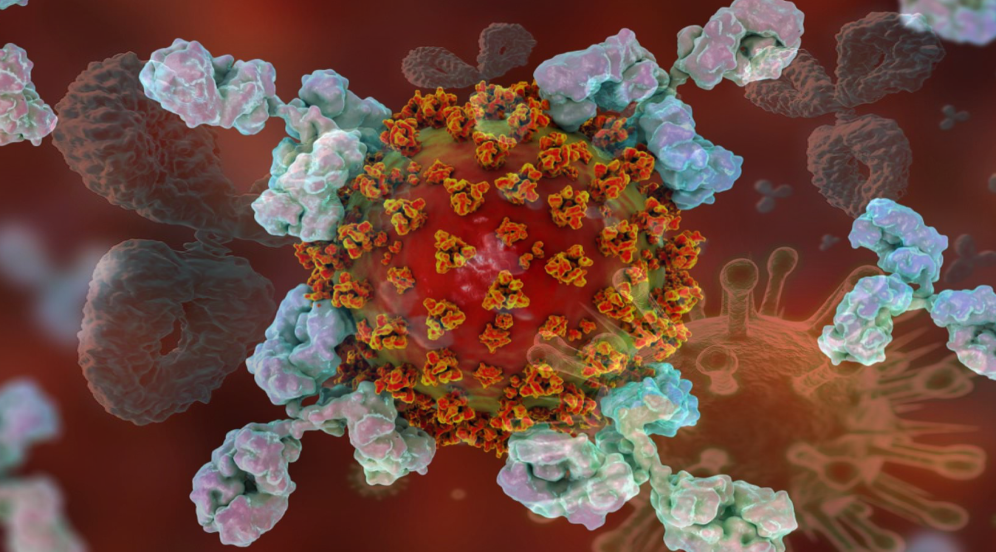
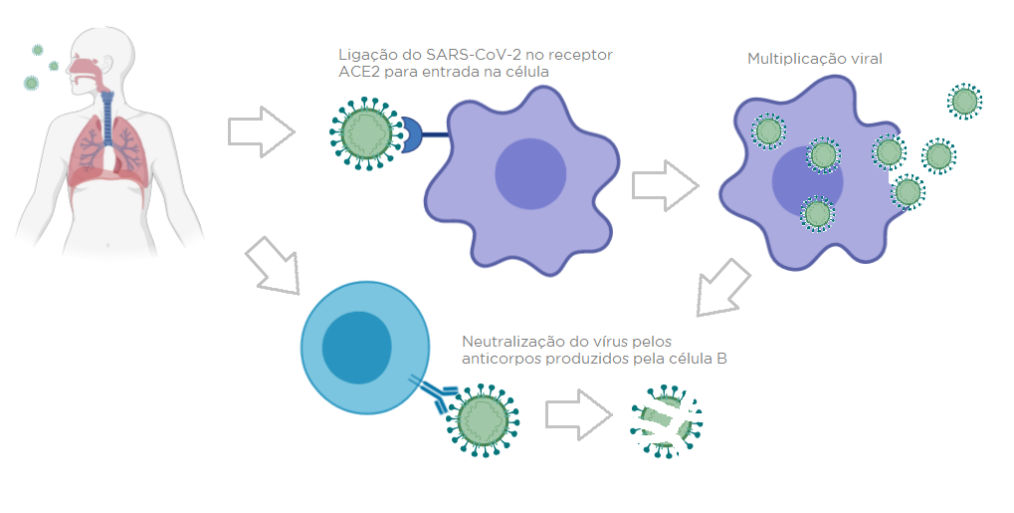
Fonte da imagem: crbm5.gov.br/imunologia-e-c…
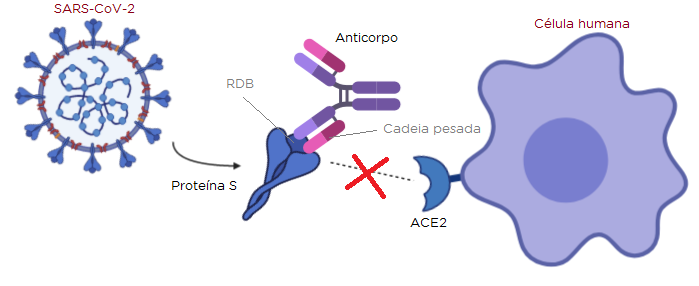
redeaanalisecovid.wordpress.com/2020/07/06/a-p…
[1] Yuan M, Liu H, Wu NC, et al. Structural basis of a shared antibody response to SARS-CoV-2 [published online ahead of print, 2020 Jul 13]. Science. 2020;eabd2321. doi:10.1126/science.abd2321
science.sciencemag.org/content/early/…
science.sciencemag.org/content/early/…
science.sciencemag.org/content/early/…
sciencedirect.com/science/articl…
sciencedirect.com/science/articl…