-iniciar su validación analítica (fase 1)
-planificar la validación clínica (fase 2)
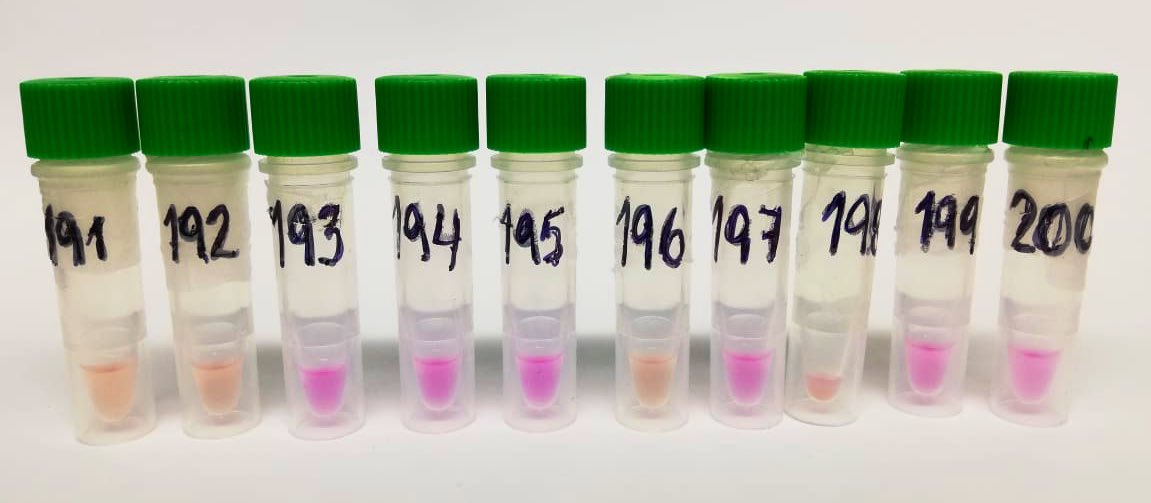
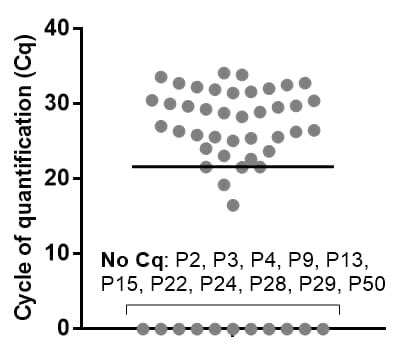
1. Concordancia total con el gold standard: 39 muestras positivas de 100 para genes virales y 98 para genes humanos (control de calidad de la muestra)
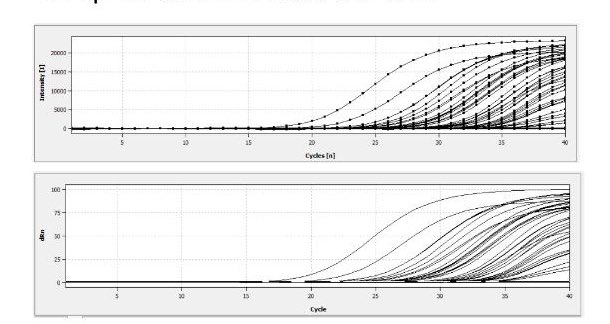
3. Límite de detección: 50 copias virales / microlitro. Detección confiable de cargas virales altas, medias y bajas