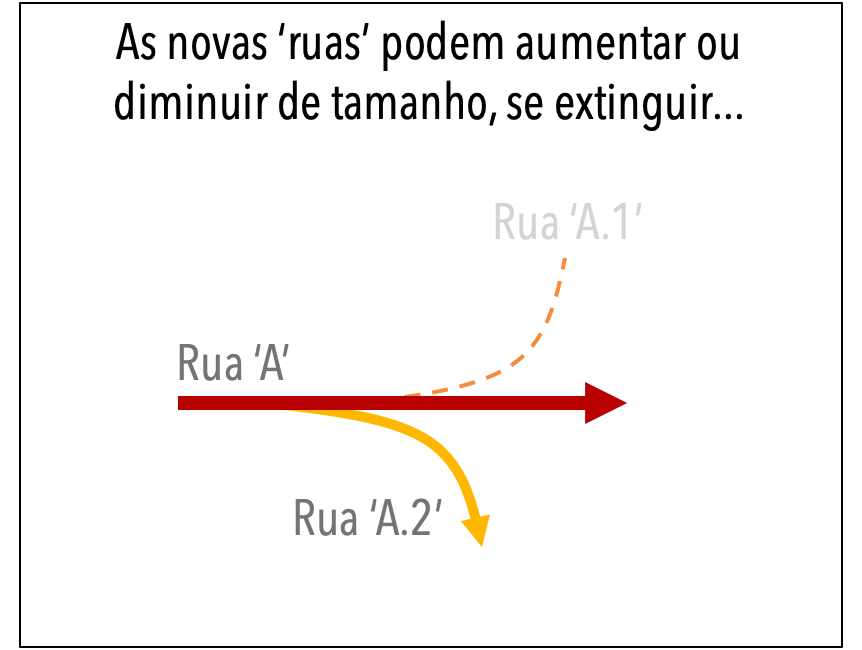
Qualquer vírus em circulação acumula mutações. A evolução nunca para. É como a construção de uma via: começa pequena, se subdivide, aumenta de tamanho… mas também diminui, e se extingue.
O sistema Pango de nomenclatura do SARS-CoV-2 visa acompanhar essas mudanças. 🧵👇
O sistema Pango de nomenclatura do SARS-CoV-2 visa acompanhar essas mudanças. 🧵👇

Quando o SARS-CoV-2 emergiu, gerando grandes surtos no início de 2020, o sistema começou classificando o vírus em duas linhagens ('ruas'): A e B.
Este artigo explica como o sistema dinâmico funciona: nature.com/articles/s4156…
Este artigo explica como o sistema dinâmico funciona: nature.com/articles/s4156…

Com mais casos de COVID-19 acontecendo, cientistas vêm sequenciando genomas do SARS-CoV-2,sempre com novas mutações que surgem naturalmente durante infecções.
Logo identificamos a expansão de certas sublinhagens, que a princípio têm o mesmo nome das linhagem da qual derivam.
Logo identificamos a expansão de certas sublinhagens, que a princípio têm o mesmo nome das linhagem da qual derivam.

A medida que mais sublinhagens (novas 'ruas') vão surgindo, em algum ponto se faz necessário atribuir novos nomes.
O time de voluntários da @PangoNetwork, do qual faço parte, fica a cargo de monitorar e nomear o surgimento de novas linhagens.
O time de voluntários da @PangoNetwork, do qual faço parte, fica a cargo de monitorar e nomear o surgimento de novas linhagens.

É comum que linhagens do SARS-CoV-2 surjam, aumentem e diminuam em frequência, até eventualmente serem tão raras, a ponto de não serem mais detectadas (podendo até se extinguir).
Usuários do sistema Pango diariamente reportam novas linhagens usando um sistema no GitHub.
Usuários do sistema Pango diariamente reportam novas linhagens usando um sistema no GitHub.
Como explico acima, a evolução do coronavírus é muito dinâmica: estamos sempre detectando novas linhagens por meio do sequenciamento genético de amostras de COVID
Mantendo um sistema de nomenclatura dinâmico, todas as linhagens são registradas neste site: cov-lineages.org
Mantendo um sistema de nomenclatura dinâmico, todas as linhagens são registradas neste site: cov-lineages.org

Sublinhagens são nomeadas em formato alfanumérico, até o quarto nível de subdivisão, quando podem receber uma nova nomenclatura em primeiro nível. Foi assim com a linhagem das variantes Delta:
B → B.1 → B.1.617 → B.1.617.2 →→→ AY
Mais informações: pango.network/new-ay-lineage…
B → B.1 → B.1.617 → B.1.617.2 →→→ AY
Mais informações: pango.network/new-ay-lineage…

Toda essa dinâmica evolutiva explica por que hoje temos tantas linhagens, como: B.1.1.7, B.1.617.2, P.1 (derivada da B.1.1.28.1).
O sistema é alfanumérico pois se adequada bem as subdivisões filogenéticas, e é neutro, para evitar estigmas ("variante indiana", "britânica", etc).
O sistema é alfanumérico pois se adequada bem as subdivisões filogenéticas, e é neutro, para evitar estigmas ("variante indiana", "britânica", etc).
Acompanhar a evolução viral de perto, e atribuir nomenclatura neutra e sistemática às novas linhagens do SARS-CoV-2, é essencial para evitar designações inadequadas, que geram confusão e ansiedade desnecessárias, como "Delta Plus", que não significa nada.
technologyreview.com/2021/08/13/103…
technologyreview.com/2021/08/13/103…
Existem no momento 3 principais sistemas de nomenclatura do SARS-CoV-2.
Pango: pango.network
Nextclade: clades.nextstrain.org
WHO: who.int/en/activities/…
Os sistemas têm similaridades, e o mesmo objetivo: nos orientar ao longo do complexo caminho evolutivo do vírus
Pango: pango.network
Nextclade: clades.nextstrain.org
WHO: who.int/en/activities/…
Os sistemas têm similaridades, e o mesmo objetivo: nos orientar ao longo do complexo caminho evolutivo do vírus

O sistema Pango é mantido por voluntários da @PangoNetwork: pango.network/committees/com…
Na matéria abaixo é possível entender como é o trabalho da rede de pesquisadores que atuam diariamente para nomear linhagens do coronavírus.
technologyreview.com/2021/07/26/103…
Na matéria abaixo é possível entender como é o trabalho da rede de pesquisadores que atuam diariamente para nomear linhagens do coronavírus.
technologyreview.com/2021/07/26/103…
Quer saber mais sobre evolução viral, mutações e novas linhagens? Explico neste outro fio.
https://twitter.com/AndersonBrito_/status/1341589500435320834
Este fio também está disponível numa versão em Inglês.
https://twitter.com/AndersonBrito_/status/1430888559150391310
• • •
Missing some Tweet in this thread? You can try to
force a refresh