Draadje.
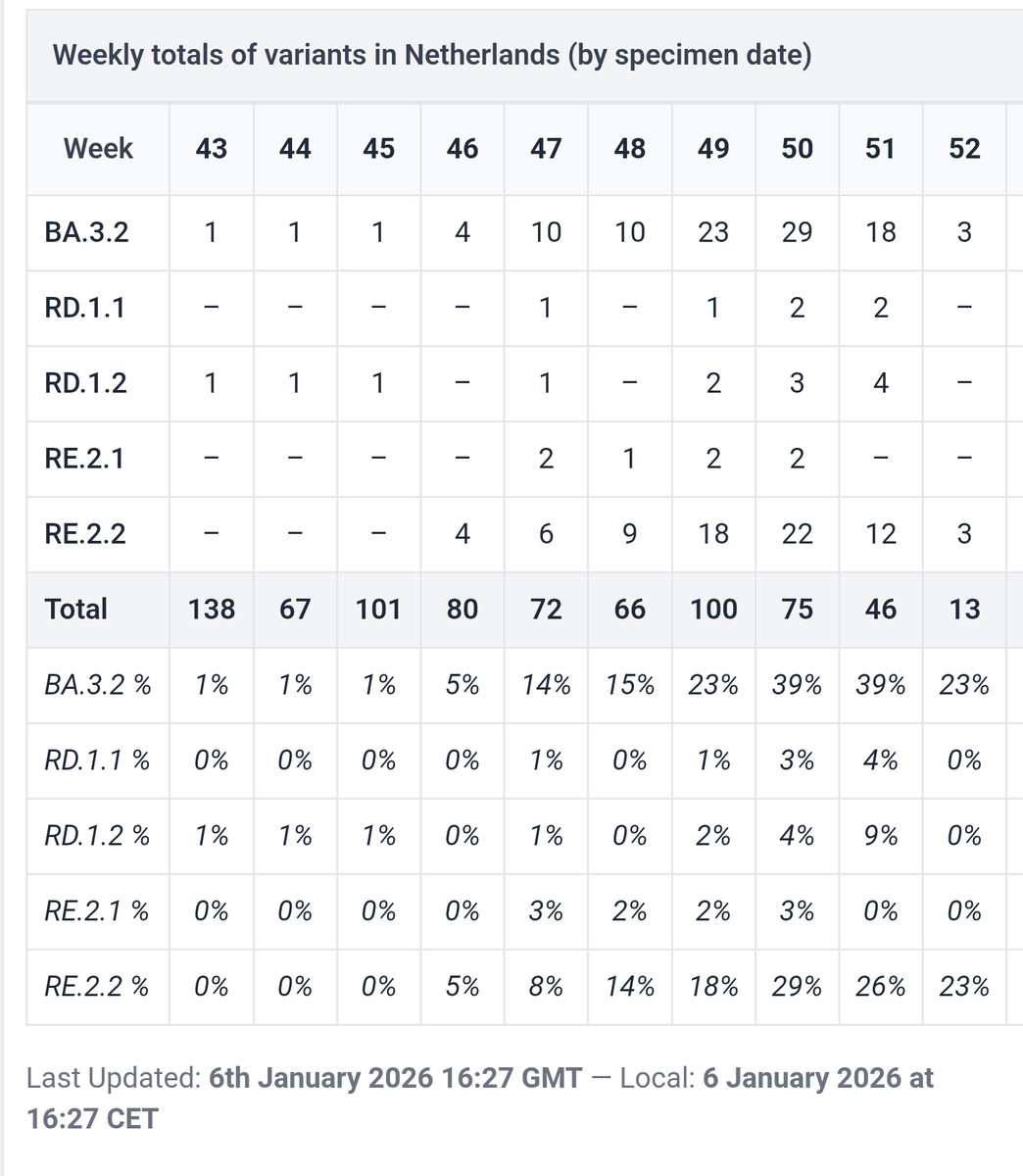
Van de laatste (+- 1000) samples die het RIVM op GISAID heeft gezet, waren er 8 AY.4.2. Daarmee komt het totaal voor Nederland op 25, 6 kwamen er uit Friesland, van 9 en 11 oktober, dus dat zou een cluster kunnen zijn. De andere 2 kwamen uit Zuid-Holland en Utrecht. >>>
Van de laatste (+- 1000) samples die het RIVM op GISAID heeft gezet, waren er 8 AY.4.2. Daarmee komt het totaal voor Nederland op 25, 6 kwamen er uit Friesland, van 9 en 11 oktober, dus dat zou een cluster kunnen zijn. De andere 2 kwamen uit Zuid-Holland en Utrecht. >>>
Delta bestaat uit B.1.617.2 en afgeleiden daarvan. Deze laatsten worden AY.x.x genoemd. AY.4.2 is dus eigenlijk B.1.617.2.4.2. Zie hier de volledige reeks Delta en nog een paar niet Delta die de laatste tijd in Nederland gevonden, gesequenced en op GISAID gezet zijn. >>> 

Geen enkele niet-Delta lijkt op te kunnen tegen Delta.
In deze selectie kun je ook zien dat B.1.617.2 nog wat harder groeit dan het gemiddelde van haar onderliggende sublineages (AY.x).
AY.9 neemt echt af, een aantal schommelen een beetje, en AY.4.2 lijkt ook hier te groeien.>>

In deze selectie kun je ook zien dat B.1.617.2 nog wat harder groeit dan het gemiddelde van haar onderliggende sublineages (AY.x).
AY.9 neemt echt af, een aantal schommelen een beetje, en AY.4.2 lijkt ook hier te groeien.>>


Ik heb hierboven week 41 eraf geknipt (waarschuwing: dat doe ik vaker!). Week 41 had namelijk 25% AY.4.2., maar dit betrof maar erg weinig samples (4/16). Dat percentage gaat dus zeker veel lager worden! Maar zijn meer dingen die meespelen. >>> 

1. Sommige regio's en labs zijn veel sneller in sequencen en hun samples op GISAID zetten dan andere en de verdeling van varianten is niet overal hetzelfde. De laatste weken kan een snelle regio dus echt zijn stempel drukken op de percentages. Dagen tussen afname en indienen: >> 

2. Het RIVM doet kiemsurveillence, wat door de aselecte steekproef een redelijk beeld geeft van de verdeling in Nederland. Niet al hun samples (wel veel) komen echter op GISAID. En hun eigen tabel maakt geen onderscheid. >>>
"Kiemsurveillance | RIVM" rivm.nl/coronavirus-co…
"Kiemsurveillance | RIVM" rivm.nl/coronavirus-co…

Daarnaast zet het RIVM en andere laberatoria ook samples op GISAID die niet bij de kiemsurveillence horen en een vertekenend beeld kunnen veroorzaken. >>>
https://twitter.com/JosetteSchoenma/status/1395758394305355776?t=_f0ajgKyM4xrosr79W1yyQ&s=19
Tegen de tijd dat een week compleet is, voor alle regio's, en een variant al redelijk vaak voorkomt, zal de invloed van een cluster bijvoorbeeld redelijk wegvallen. Maar als de helft van de samples van een bepaalde variant in 1 week een cluster was, dan vertekend dat. >>>
Terug naar AY.4.2. In de UK houden ze hem (niet voor niks) goed in de gaten. Zie hier hun laatste rapport (PHE). Zo fijn dat zij zoveel meteen en delen!
assets.publishing.service.gov.uk/government/upl…
assets.publishing.service.gov.uk/government/upl…
Zij hebben de AY.4.2 uitgeroepen tot Variant Under Investigation en noemen hem VUI-21OCT-01.
Hij groeit laatste tijd 17% harder dan de rest van de varianten.
En het % van contacten dat aangestoken wordt (SAR) ligt iets hoger. Dat verschil is binnen huishoudens significant. >>>


Hij groeit laatste tijd 17% harder dan de rest van de varianten.
En het % van contacten dat aangestoken wordt (SAR) ligt iets hoger. Dat verschil is binnen huishoudens significant. >>>



Waar ik wel (hopelijk onterecht!) van schrok, is dat het percentage van mensen besmet met de AY.4.2. iets vaker bij de EHBO kwamen of opgenomen werden en iets vaker overleden. Maar de aantallen zijn nog te laag en de onzekerheidsmarges te groot voor verregaande conclusies. >>> 



Dit zette me echter wel aan het denken. Wat nou als deze variant extra goed mensen kan infecteren die Astrazeneca gekregen hebben en dat dit vaccin ook minder goed beschermd tegen ernstige ziekte. Dan zou het effect in landen met minder AZ en meer Pfizer anders kunnen zijn. 🤔 >>
Toen ben ik naar de leeftijdsverdeling gaan kijken. 5 van de 23 AY.4.2 samples waarvan de leeftijd bekend was, betrof 60-65 jarigen, die vaak Astrazeneca hebben gekregen. Dat lijkt me veel in verhouding met alle samples. Maar goed, veel te weinig samples en veel te voorbarig! >>> 

Maar misschien wel iets om in het achterhoofd te houden en te blijven monitoren. Ook qua ziekenhuisopnames etc..
Ik vind ook dat mensen na Janssen allang een mRNA booster hadden moeten krijgen en na Astrazeneca is dat misschien ook niet zo'n slecht idee. Ook voor zorgmedewerkers!
Ik vind ook dat mensen na Janssen allang een mRNA booster hadden moeten krijgen en na Astrazeneca is dat misschien ook niet zo'n slecht idee. Ook voor zorgmedewerkers!
• • •
Missing some Tweet in this thread? You can try to
force a refresh