
How to get URL link on X (Twitter) App

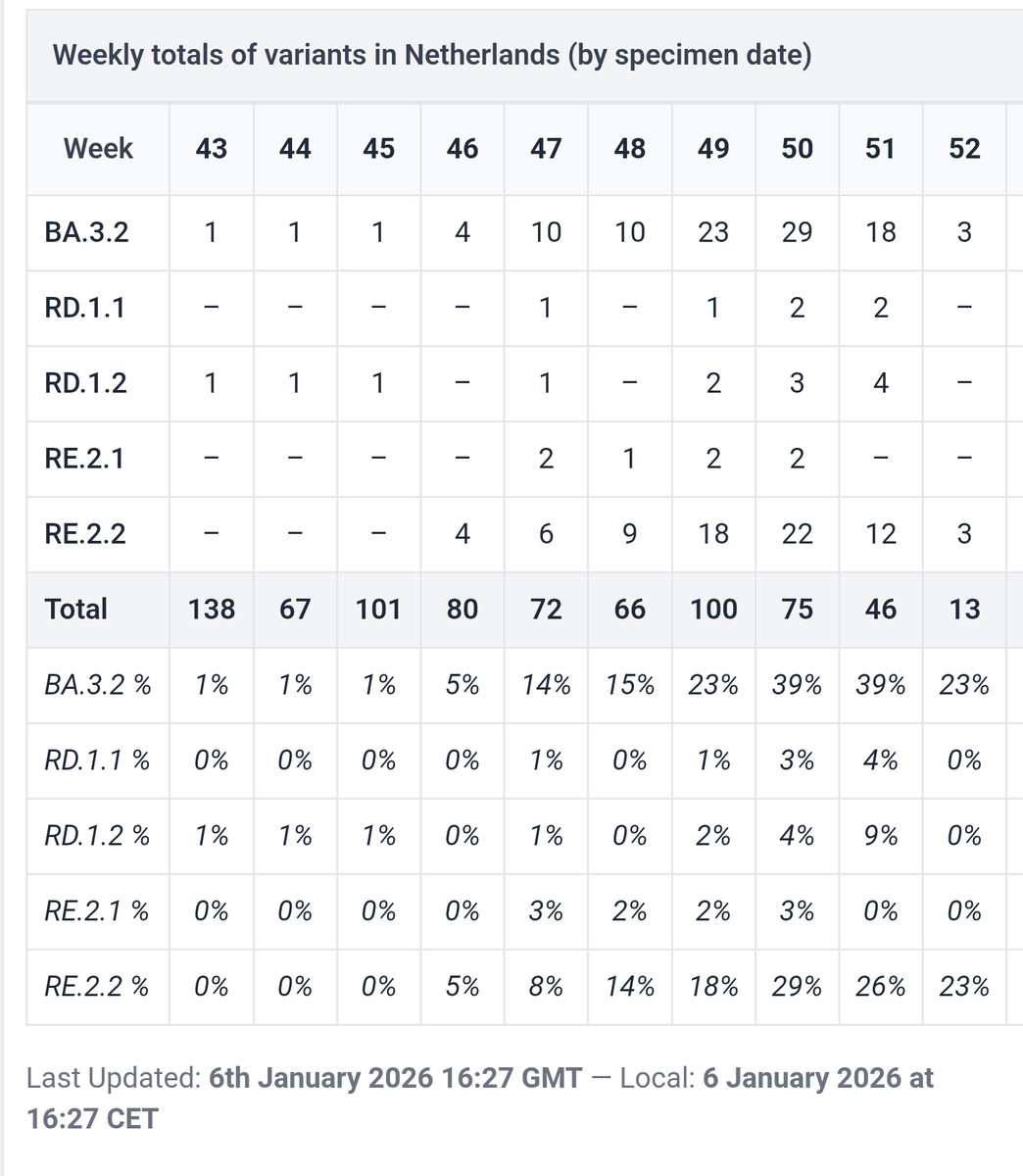
 Het SC2 niveau op het eind van 2025 was, voor zover bekend, nog niet aan het stijgen in het rioolwater. Dit was ook niet te verwachten, aangezien BA 3.2* toen waarschijnlijk ook nog niet boven de 50% zat
Het SC2 niveau op het eind van 2025 was, voor zover bekend, nog niet aan het stijgen in het rioolwater. Dit was ook niet te verwachten, aangezien BA 3.2* toen waarschijnlijk ook nog niet boven de 50% zat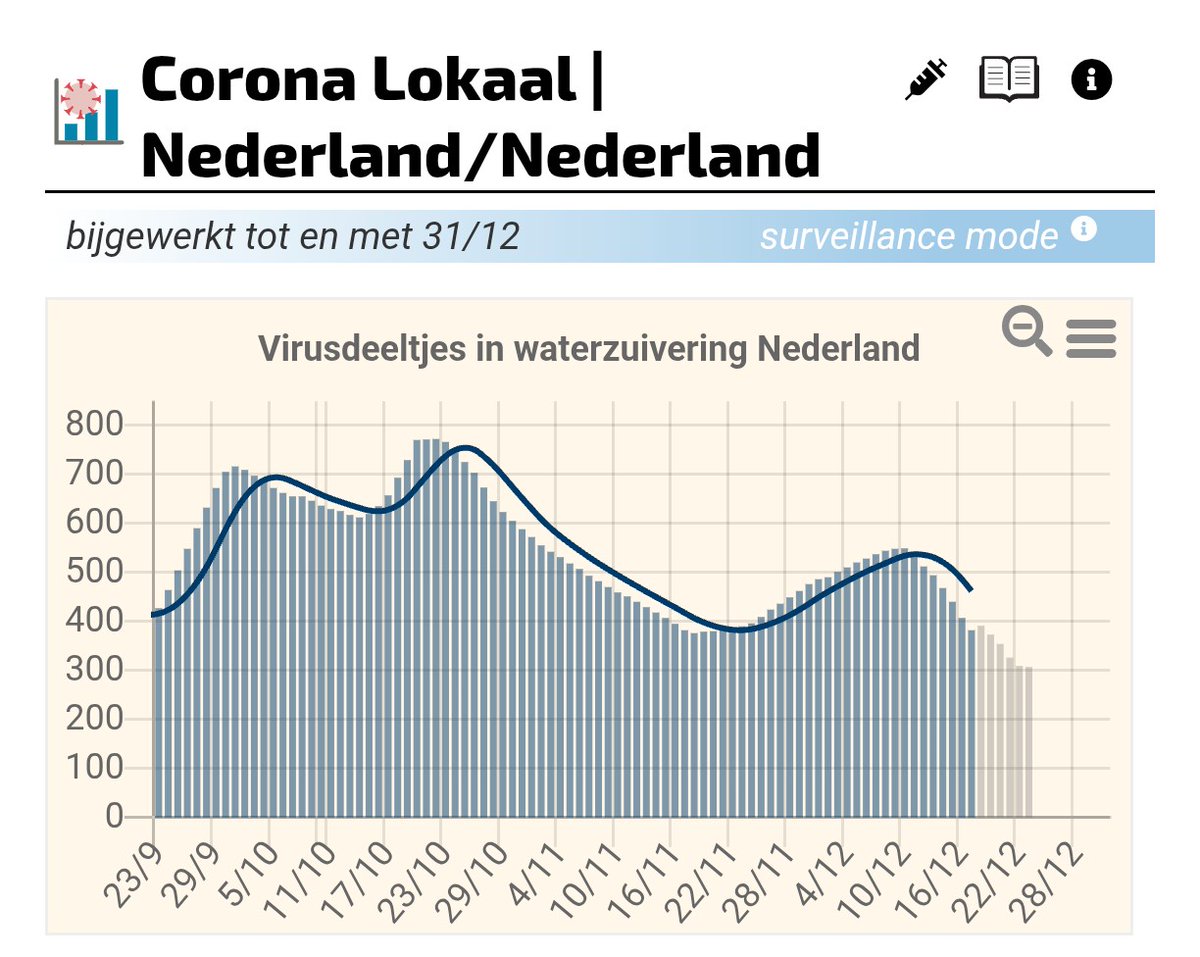

 XBF laat al een tijd een flinke groei zien in de wereld.
XBF laat al een tijd een flinke groei zien in de wereld.




 Ik heb CH.1.1* en XBB.1.5* nog even los in een logaritmische schaal gegoten, om te kunnen zien welke het hardst stijgt.
Ik heb CH.1.1* en XBB.1.5* nog even los in een logaritmische schaal gegoten, om te kunnen zien welke het hardst stijgt.

 Ook in de landelijke surveillence is de XBB.1.5 in opkomst, maar het beeld is daar minder duidelijk.
Ook in de landelijke surveillence is de XBB.1.5 in opkomst, maar het beeld is daar minder duidelijk.

 CH.1.1* has the highest percentage of those. That makes sense, since it's very common in the UK.
CH.1.1* has the highest percentage of those. That makes sense, since it's very common in the UK.



https://twitter.com/JosetteSchoenma/status/1611312032778706945
 Ook in de inschatting voor de toekomst staan CH.1.1, XBB.1.5 en XBF nog niet, dus die mag ook 🚮.
Ook in de inschatting voor de toekomst staan CH.1.1, XBB.1.5 en XBF nog niet, dus die mag ook 🚮.


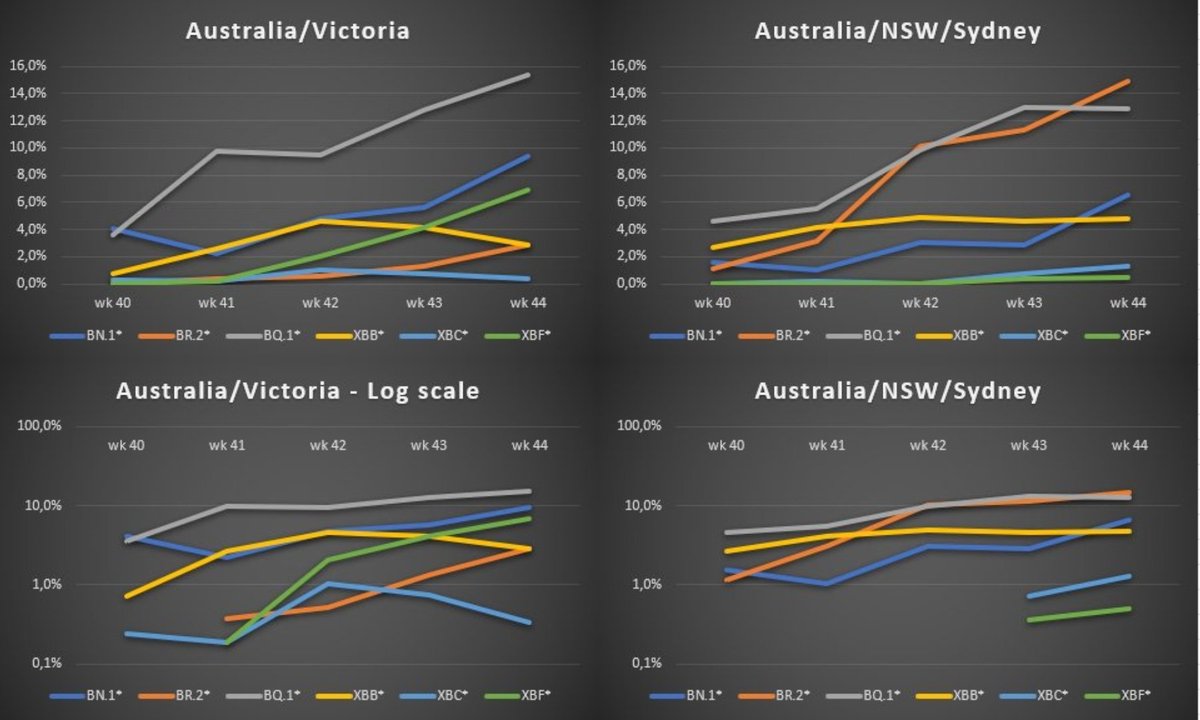
 I didn't make graphs for Australia for a while, because there is bias in the data, because certain samples are prioritised for sequencing. Like samples of people that are hospitilised, from travellers and SGTP samples. At least in NSW.
I didn't make graphs for Australia for a while, because there is bias in the data, because certain samples are prioritised for sequencing. Like samples of people that are hospitilised, from travellers and SGTP samples. At least in NSW. 


 Op log-schaal kun je zien dat BQ.1.1* ietsje harder groeit dan BQ.1* en dat XBB de laatste week ook flink gegroeid is. Maar die lijn is niet vloeiend en de aantallen zijn nog klein. Even afwachten nog wat dat gaat worden.
Op log-schaal kun je zien dat BQ.1.1* ietsje harder groeit dan BQ.1* en dat XBB de laatste week ook flink gegroeid is. Maar die lijn is niet vloeiend en de aantallen zijn nog klein. Even afwachten nog wat dat gaat worden. 

https://twitter.com/JosetteSchoenma/status/1577377625743532032
 Maar ik herhaal nogmaals dat ik denk dat een BA.1 vaccin nemen beter is dan helemaal geen vaccin nemen.
Maar ik herhaal nogmaals dat ik denk dat een BA.1 vaccin nemen beter is dan helemaal geen vaccin nemen.


 - cases are often only rising once new variants become dominant, which might not be the case yet in several regions.
- cases are often only rising once new variants become dominant, which might not be the case yet in several regions.


https://twitter.com/Viroloog/status/1531167639799746561In the Netherlands, in data from random surveillence (2 weeks behind on ☝️) BA.2.12.1 has the most samples of the 3 main growing variants recently, but BA.5 seems to grow a littlebit faster.


https://twitter.com/Viroloog/status/1531167639799746561Dit is het beeld in de kiemsurveillence (random) voor Nederland (tot aan week 19 i.p.v. 21).





https://twitter.com/CzerwinsMarcin/status/1502707012089561094

 Er zijn al eerder onderzoeken geweest met een vergelijkbaar resultaat. AA zijn hier FUT2 non secretors (niet uitscheiden), en AG en GG secretors.
Er zijn al eerder onderzoeken geweest met een vergelijkbaar resultaat. AA zijn hier FUT2 non secretors (niet uitscheiden), en AG en GG secretors. https://twitter.com/CzerwinsMarcin/status/1502728875377311754?t=GYAf2QHDP70e3RdirxJ-oQ&s=19


 Japan has a good border surveillence system and publishes the countries the travelers went to and which variant they carried.
Japan has a good border surveillence system and publishes the countries the travelers went to and which variant they carried.https://twitter.com/KawasakiKR11/status/1484351931904454664?t=uhLdhs4_UsuV8LtVT6k4-A&s=19

https://twitter.com/c_reusken/status/1473577227715584008SGTF = S Gene Target Failure


 Omicron is inmiddels in alle provincies, behalve Drenthe en Friesland, gevonden. In provincies met veel inwoners worden vaak ook meer samples gesequenced, dus dat levert natuurlijk bias op.
Omicron is inmiddels in alle provincies, behalve Drenthe en Friesland, gevonden. In provincies met veel inwoners worden vaak ook meer samples gesequenced, dus dat levert natuurlijk bias op.


https://twitter.com/CorneliusRoemer/status/1470515881692254218?t=tXlEGCyRK4VXadVK9Fg0Cg&s=19




 Denemarken lijkt een beetje een vreemde eend in de bijt, maar ik verwacht dat ze daar wat clusters gesequenced hebben, toen de AY.4.2 net op kwam. >>>
Denemarken lijkt een beetje een vreemde eend in de bijt, maar ik verwacht dat ze daar wat clusters gesequenced hebben, toen de AY.4.2 net op kwam. >>> 

https://twitter.com/JosetteSchoenma/status/1452242185005539332

 De SAR blijft wel nog steeds hoger voor AY.4.2 dan voor Delta.
De SAR blijft wel nog steeds hoger voor AY.4.2 dan voor Delta.https://twitter.com/JosetteSchoenma/status/1452240663634923535?t=eR8NW4yNNJxY85uBG8U5UA&s=19
