Hace dos años propusimos un plan para crear una red de vigilancia genómica de SARS-CoV-2 en Perú. Este mes cerramos el proyecto y agotamos los últimos recursos disponibles. Va un resumen del trabajo y lecciones aprendidas en dos años de estudiar genomas en pandemia. 🧵
1/24

1/24


Este post se basa en nuestra presentación hace unas semanas en el evento organizado por @Concytec y @UKinPeru. Aquí la nota de prensa.
gob.pe/institucion/co…
2/
gob.pe/institucion/co…
2/
Empezamos en abril 2020 con la primera ronda de proyectos de emergencia COVID-19 del @Concytec. En la primera fase, optimizamos métodos para secuenciar el genoma de SC-2 y estudiar su evolución local. La meta fue generar 1,000 genomas virales en 2020.
3/
fondecyt.gob.pe/convocatorias/…
3/
fondecyt.gob.pe/convocatorias/…
En mayo, INS había reportado el primer genoma en Perú e iniciaba su propia estrategia de vigilancia genómica.
4/
doi.org/10.1128/MRA.00…

4/
doi.org/10.1128/MRA.00…


En julio, al fin obtuvimos todos los reactivos y permisos para iniciar. Los primeros análisis confirmaron la introducción del SC-2 desde Europa y la dominancia temprana del linaje B.1.1.1, que meses después evolucionaría hacia una forma más contagiosa.
5/
5/
https://twitter.com/pablotsukayama/status/1313990076993605633
Cerramos 2020 con 1200+ genomas entre INS+UPCH y noticias de variantes más transmisibles desde Inglaterra, Sudáfrica y Brasil. Cerraba el proyecto y nos quedamos sin fondos cuando más necesitábamos monitorear la entrada de nuevas variantes en el país.
6/
larepublica.pe/domingo/2021/0…
6/
larepublica.pe/domingo/2021/0…
En marzo 2021, obtuvimos S/800,000 adicionales de Concytec para escalar y descentralizar nuestra estrategia de vigilancia. Se unían nuevos colaboradores de UNSA, UNTRM y UNMSM.
7/
7/
https://twitter.com/pablotsukayama/status/1371438355083313152
Por entonces, llegábamos al pico de la segunda ola en Perú y la cosa pintaba muy mal. Se pensaba que la variante Gamma, importada desde Brasil, sería responsable de la nueva ola de contagios.
8/

8/

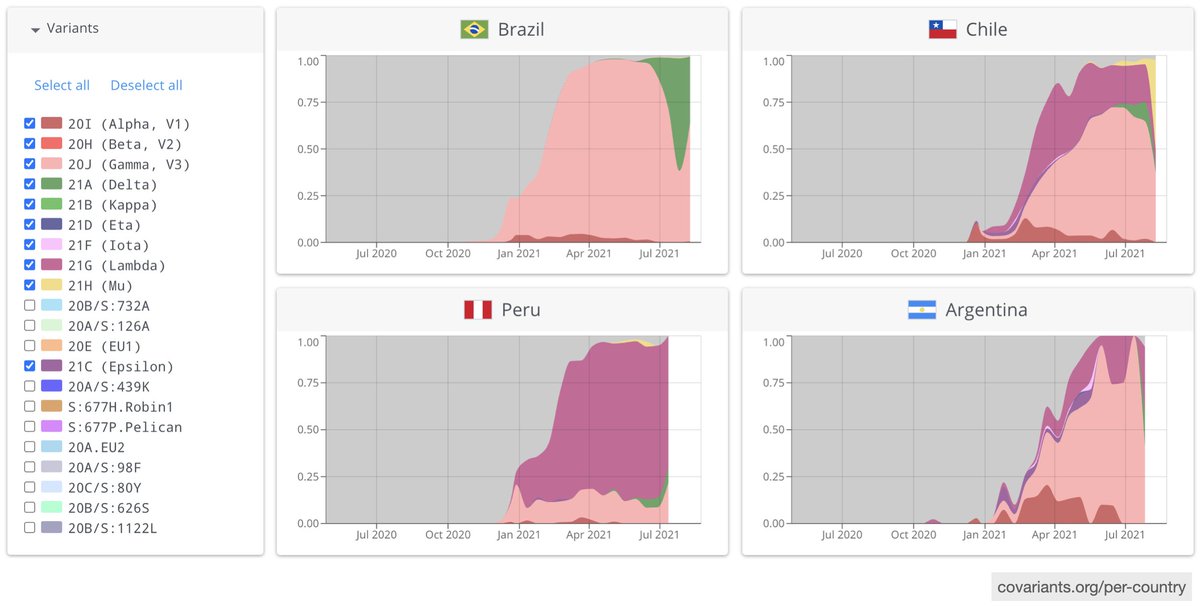
En abril 2021, reportamos un nuevo linaje, C.37, que se expandía rápidamente en Peru, Chile y Argentina y competía con Gamma en la región. Inicialmente se le llamó ‘Variante Andina’.
9/
9/
https://twitter.com/pablotsukayama/status/1386054160378912769
Al comienzo, INS minimizó los resultados. En mayo, luego escalar sus sistema de vigilancia a nivel nacional, empezaron a generar 1000+ genomas por mes y confirmaron la presencia de C.37 en 70%+ de sus muestras.
10/
web.ins.gob.pe/es/prensa/noti…
web.ins.gob.pe/index.php/es/p…
10/
web.ins.gob.pe/es/prensa/noti…
web.ins.gob.pe/index.php/es/p…

En junio 2021, luego de varias discusiones con expertos en la región, OMS designó a C.37 como ‘Variante de Interés Lambda’ 6+ meses después de haberse identificado por primera vez y luego exportarse a 30+ países.
11/
who.int/en/activities/…

11/
who.int/en/activities/…


(El paper sobre Lambda, ya revisado por pares, fue publicado en Octubre en el journal Microbiology Spectrum de @ASMicrobiology)
12/
doi.org/10.1128/Spectr…
12/
doi.org/10.1128/Spectr…

En mayo, llegarían los primeros casos de Delta a Perú, que reemplazaría lentamente a Lambda, Gamma y Mu hasta volverse la variante dominante en la región a finales de 2021.
13/
13/
https://twitter.com/pablotsukayama/status/1462868895757385737
En agosto, ya con los casos de bajada, pudimos completar la transferencia de protocolos a nuestros amigos de UNSA en Arequipa, generando las primeras secuencias fuera de Lima.
14/
14/
https://twitter.com/pablotsukayama/status/1429107330612928512
En octubre, terminamos de implementar los métodos en Chachapoyas, gracias a la colaboración con UNTRM y DIRESA Amazonas.
15/
15/
https://twitter.com/pablotsukayama/status/1451916492069646343
En noviembre aparece Omicron. Su rápida identificación es posible gracias al excelente trabajo de científicos en Sudáfrica. El mundo les responde con cierre de vuelos que no funcionaron: Omicron llegaría a todos los países para enero 2022.
16/

16/


En febrero, nuestros colaboradores en San Marcos empiezan a generar nuevos datos de vigilancia en Lima y reportamos los primeros casos de Omicron BA.2 en Perú junto a INS.
17/
gestion.pe/peru/covid-19-…
17/
gestion.pe/peru/covid-19-…
Marzo 2022: Entre UPCH, PUCP, UNMSM, UNSA, UNTRM logramos descentralizar la vigilancia genómica y aportamos 2500+ genomas de casos en Amazonas, Arequipa, Cusco, Lima y Callao, que representan el 12% de los casi 20,000 genomas peruanos reportados en GISAID desde 2020.
18/
18/

No hemos podido conseguir financiamiento para mantener la red en funcionamiento y ahora toda la vigilancia de SC-2 queda en manos de INS. Esperemos que MINSA y autoridades sigan financiando este programa y que aprecien el aporte de las universidades en los dos últimos años.
19/
19/
Más allá del Covid, esta red de universidades y laboratorios regionales puede ayudar ante la amenaza de nuevas pandemias. Respondimos muy lento frente el Covid y debemos ser más eficientes ante futuros virus de potencial pandémico.
20/
technologyreview.com/2022/02/23/104…
20/
technologyreview.com/2022/02/23/104…
OMS acaba de lanzar una estrategia global de 10 años para la vigilancia genómica de patógenos, enfatizando la necesidad de desarrollar capacidades locales y promover #datosabiertos para mejorar la capacidad de respuesta de todos los países.
21/
who.int/news/item/30-0…



21/
who.int/news/item/30-0…




La pandemia no ha terminado. Llegarán nuevas olas y variantes mientras el virus se siga transmitiendo y no haya una distribución justa de vacunas a nivel global. Debemos seguir rastreando la evolución del virus. No es momento de bajar la guardia.
22/
22/
Todo nuestro agradecimiento a las personas e instituciones que hicieron posible este trabajo. Tenemos una red sólida de científicos peruanos que aún tiene mucho por aportar. Si conocen de fundaciones o empresas que quieran financiar nuestro trabajo, ¡pasen la voz!
23/
23/

Y un agradecimiento especial a este maravilloso grupo de jóvenes científicxs que tengo el honor de dirigir en @CayetanoHeredia. Nada de esto sería posible sin ellos. Seguiremos informando🖖
24/
Fin.
24/
Fin.

¡Muchas gracias por todo el apoyo! Una de las cosas más bonitas en estos dos años ha sido conocer a tanta gente nueva y poder mostrar que científicxs + universidades podemos aportar desde lo nuestro.
Talento y ganas, hay. Pero aún falta bastante apoyo.
Talento y ganas, hay. Pero aún falta bastante apoyo.
Recuerdo que hace un año varios candidatos a presidente y congreso hablaban de apoyar a los científicos locales. Y que la toma de decisiones para el manejo de la pandemia se basaría en evidencia científica. ¿Qué fue de ellos?
• • •
Missing some Tweet in this thread? You can try to
force a refresh