hautconseildesbiotechnologies.fr/sites/www.haut…
(et bon je parle pas des cas ou ya carrément pas de peer review, ou qu'il est totalement baclé)
C'est ce qui a amené les chercheurs, notamment John Ioannidis, à dire que la plupart des résultats positifs publiés sont faux.
journals.plos.org/plosmedicine/a…
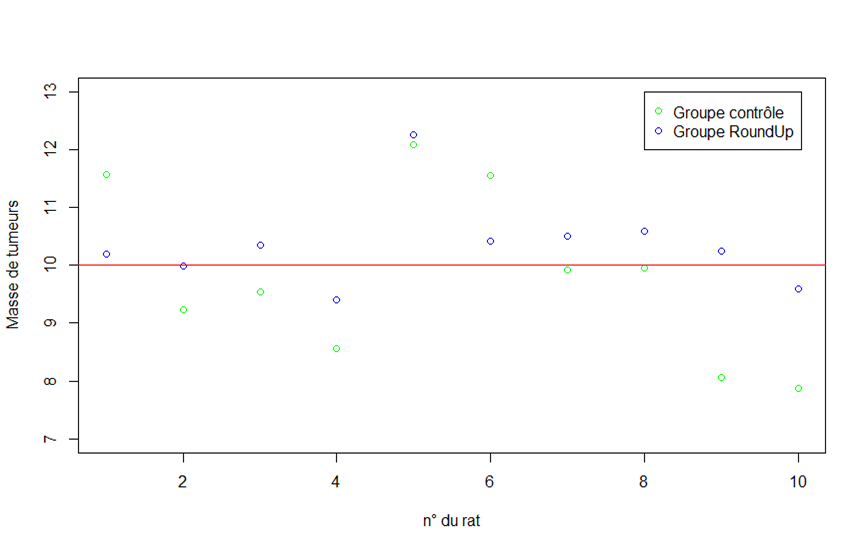
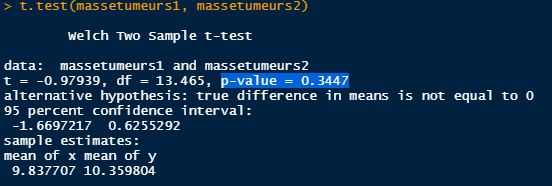
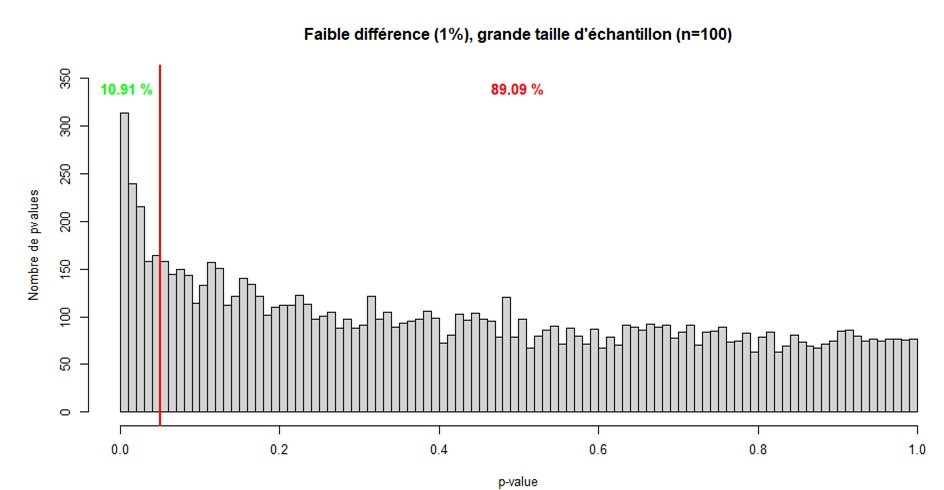
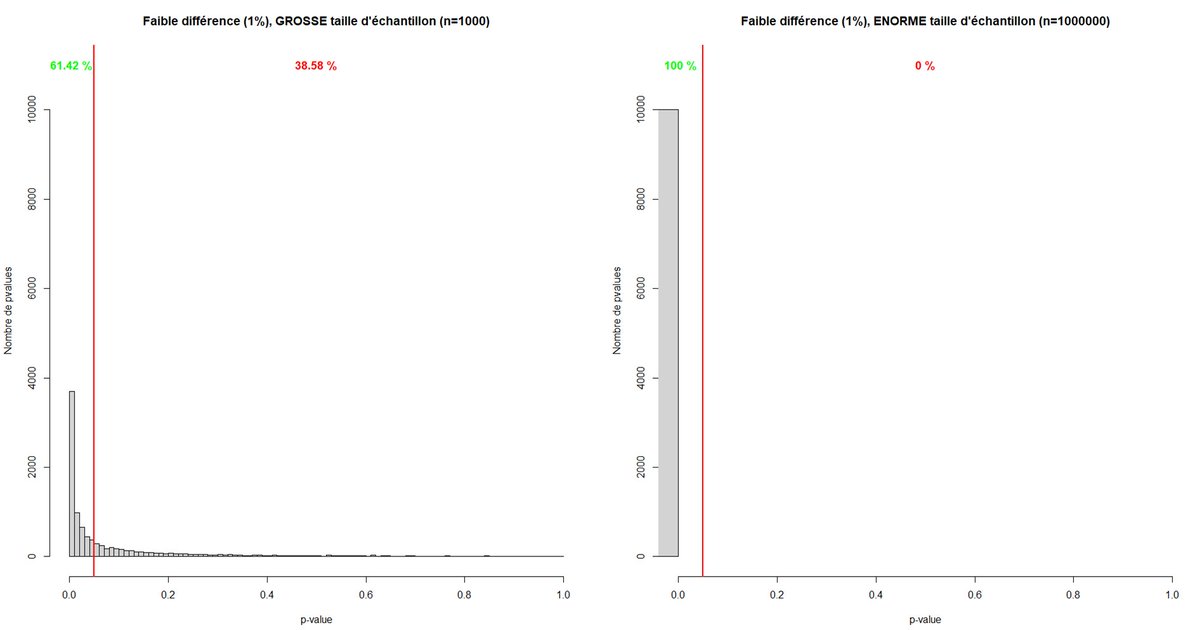
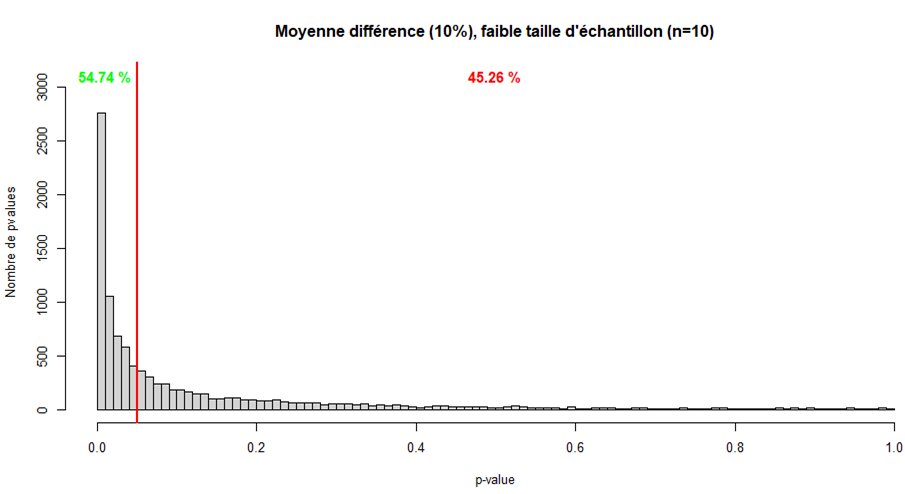
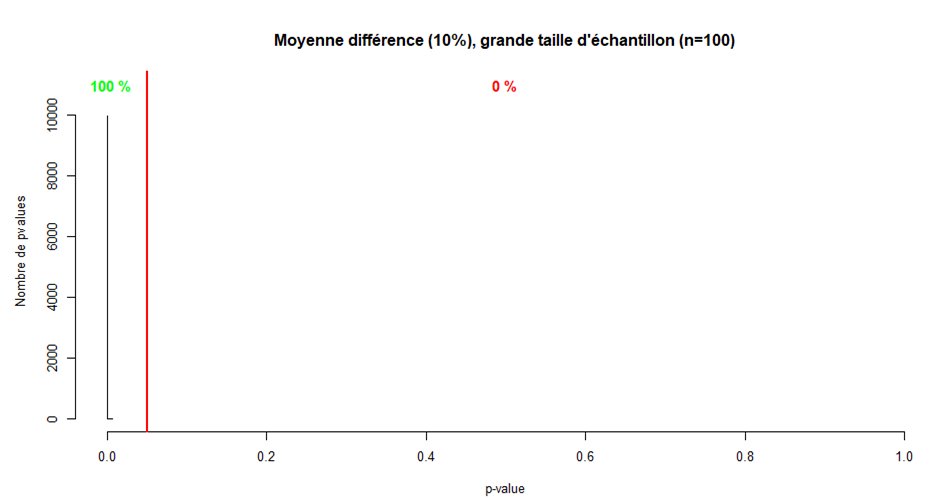
La taille d'échantillon n'influe en rien sur l'importance de l'effet, et vice versa.
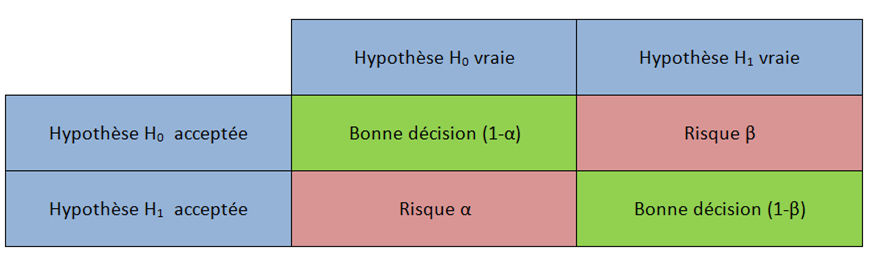
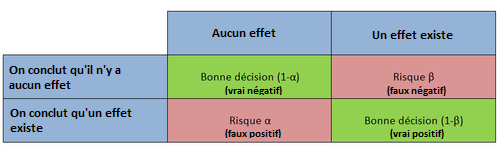
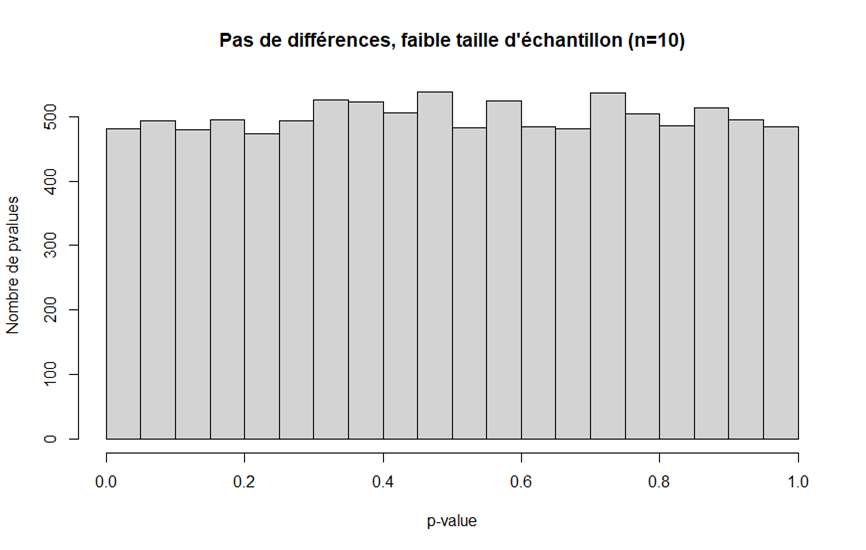
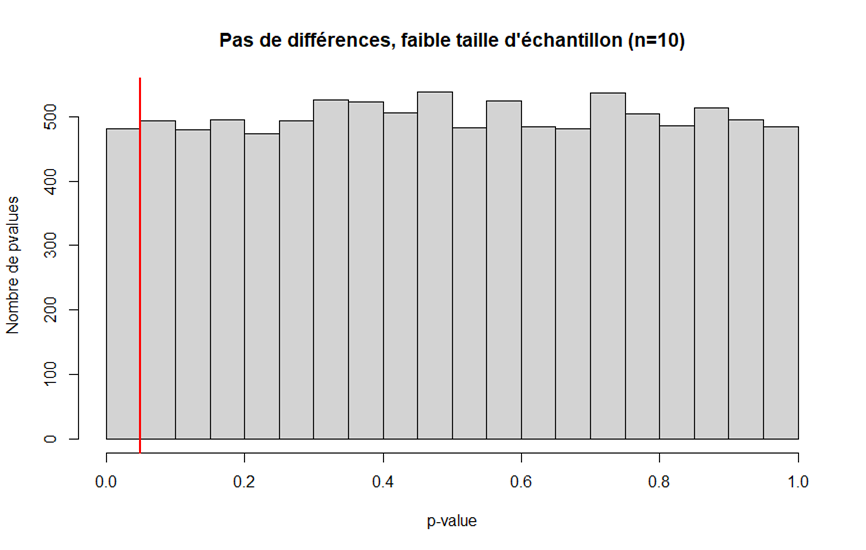
- le risque alpha ne dépend pas de la taille d'échantillon
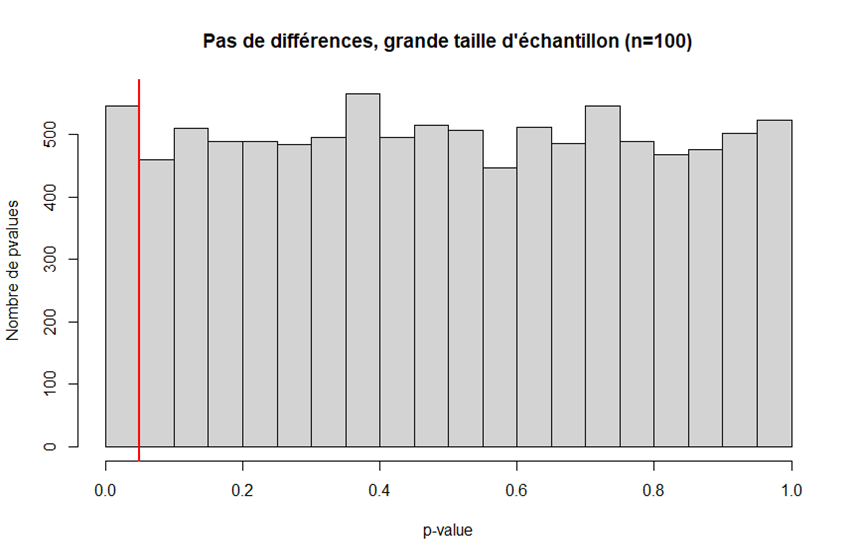
- un grand échantillon permet de diminuer le risque beta et donc plus facilement déceler un effet réel
drive.google.com/file/d/1-uYUyR…