
A 2+ meses de iniciar el proyecto de vigilancia genómica, el equipo en @CayetanoHeredia ha secuenciado 131 genomas peruanos de SARS-CoV-2 de marzo-agosto 2020. Junto a 146 secuencias del @INS_PERU nos permiten entender los eventos iniciales de la pandemia en Perú. 1/
El proyecto inició como una colaboración con @PUCP e INS. Ahora se suman @UNMSM_, @UNSA_Oficial de Arequipa, @untrm de Chachapoyas, @tumigenomics y el @sangerinstitute de Inglaterra. Financian @FondecytPeru, @VLIRUOS y @IDB_Lab 2/ 

El trabajo se dividió en tres equipos: (1) Procesamiento y extracción de ARN de muestras que llegan de INS, en un laboratorio BSL2+ acondicionado y certificado (en colaboración con @EmergeUpch). 3/ 



(2) Conversión a cDNA, amplificación del genoma viral (utilizando una adaptación del protocolo @NetworkArtic), preparación de librerías genómicas y secuenciamiento en instrumentos #Illumina en la Unidad de Epimiología Molecular de @TropicalesUPCH 4/ 







También trabajamos con el laboratorio de @MarianaLeguia en @pucp en la optimización del protocolo de secuenciamiento y la cuantificación de nuestras librerías Gracias Mariana y Alejandra! 5/ 







(3) Procesamiento de secuencias y análisis bioinformático. @quipupe et al han creado un pipeline para control de calidad, mapeo al genoma de referencia, ensamblaje de novo y búsqueda de errores antes de subir los datos a GISAID (ensamblajes) y a Genbank (datos crudos). 6/ 

Aquí pueden acceder a un tutorial de este pipeline para que puedan replicar el análisis: github.com/quipupe/from-a… 7/
Nos tomó varias semanas (y reactivos) optimizar protocolos para obtener secuencias de alta calidad. Hace un mes (10-sep) empezamos a subir los primeros genomas a @GISAID. La semana pasada cargamos 46, de casos de fines de agosto 2020. 8/
[
[
https://twitter.com/nextstrain/status/1313156030579257347]
Para poner el número en contexto: al día de hoy se han generado 140,000 genomas de SARS-CoV-2 en laboratorios de más de 100 países. Nunca antes se había generado tanta información genómica de un organismo en tan poco tiempo. 9/ 
Sin embargo, solo ∼2,000 de esos 140,000 genomas (menos del 2%) provienen de Sudamérica, a pesar de reportar el 22% de los casos a nivel mundial. 10/
Aún así, esta información ya nos permite estudiar la transmisión del virus a escala mundial, como pueden ver en esta excelente animación de @evogytis, un mago de #Matplotlib. 11/
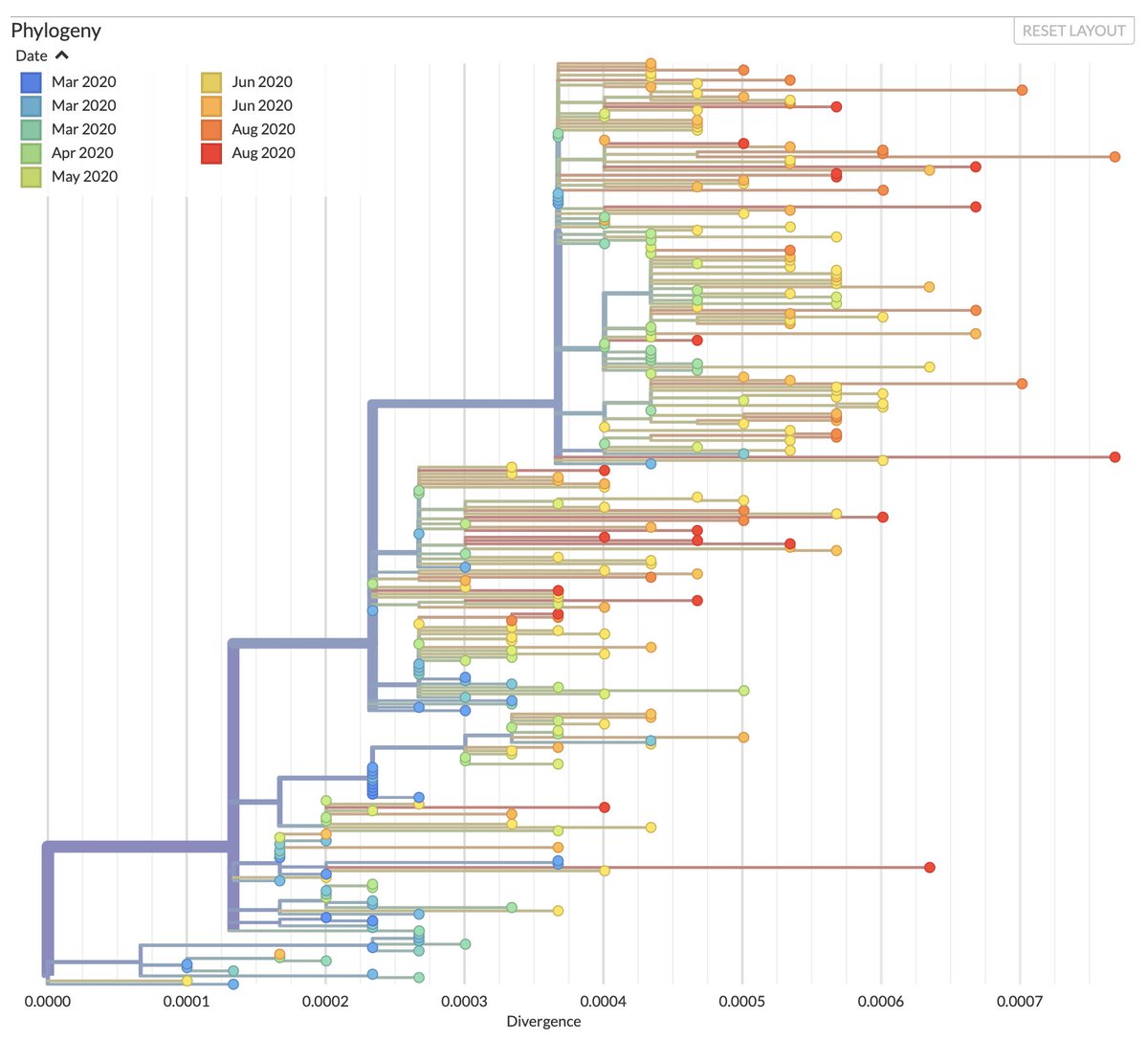
Estos son los 277 genomas que hemos usado para nuestro análisis, ordenados por linaje (Pangolin), en un árbol filogenético y organizados por región y fecha de muestreo. Les invito a que exploren los datos en este enlace: [nextstrain.org/community/quip…] 12/ 

(aquí un tutorial que explica como leer e interpretar estos árboles filogenéticos.) 13/ neherlab.org/201901_krisp_a…
Vemos también que los muestreos de INS y UPCH se complementan entre sí y nos permiten estudiar las primeras introducciones del coronavirus en Perú + las cadenas de transmisión que se han mantenido hasta agosto. 14/ 

Los colegas del INS se han enfocado en las muestras de marzo-abril, publicando recientemente DOS preprints que describen sus resultados. 15/
Interesante que sean de grupos distintos de la misma institución, trabajando sets distintos de muestras, reportando resultados similares de forma independiente. 16/ biorxiv.org/content/10.110…
El segundo grupo (que publicó el 16-set) incorpora en su análisis los 149 genomas del primer grupo (sin citar a los otros autores) y sube sus muestras a GISAID recién después de publicar su preprint. ¿Qué onda, INS? Not cool. 17/ biorxiv.org/content/10.110…
Además, vemos que recientemente (22-set) subieron 87 nuevos genomas a GISAID (¡bien!) pero sin información de región, sexo o edad del paciente (¡mal!). 80% de secuencias en GISAID contiene esta metadata y sin ella no podemos analizar los datos de manera apropiada. 18/ 



Pero bueeeeeeeno... ¿Qué sabemos sobre el inicio de la pandemia en Perú? (1) Tuvimos múltiples introducciones del virus en marzo y (2) estas generaron múltiples cadenas de transmisión local. 19/
O sea: tuvimos varios "pacientes cero" y varias de estas "chispas" generaron suficientes contagios para iniciar el incendio que es hoy la pandemia en Perú. 20/ 

Esta historia se repite en varios otros países. Por ejemplo, Brasil reporta más de 100 introducciones entre febrero y marzo. Se estiman más de 1,300 introducciones en el Reino Unido. Reflejo de un mundo altamente conectado. 21/
[science.sciencemag.org/content/369/65…]
[science.sciencemag.org/content/369/65…]
¿Qué más? Sabemos que el virus sigue mutando, a un promedio de 2 mutaciones por mes. Podemos observar esto en los genomas de junio y agosto, que presentan la mayor cantidad de SNPs (mutaciones) con respecto al virus original en Wuhan. 22/ 

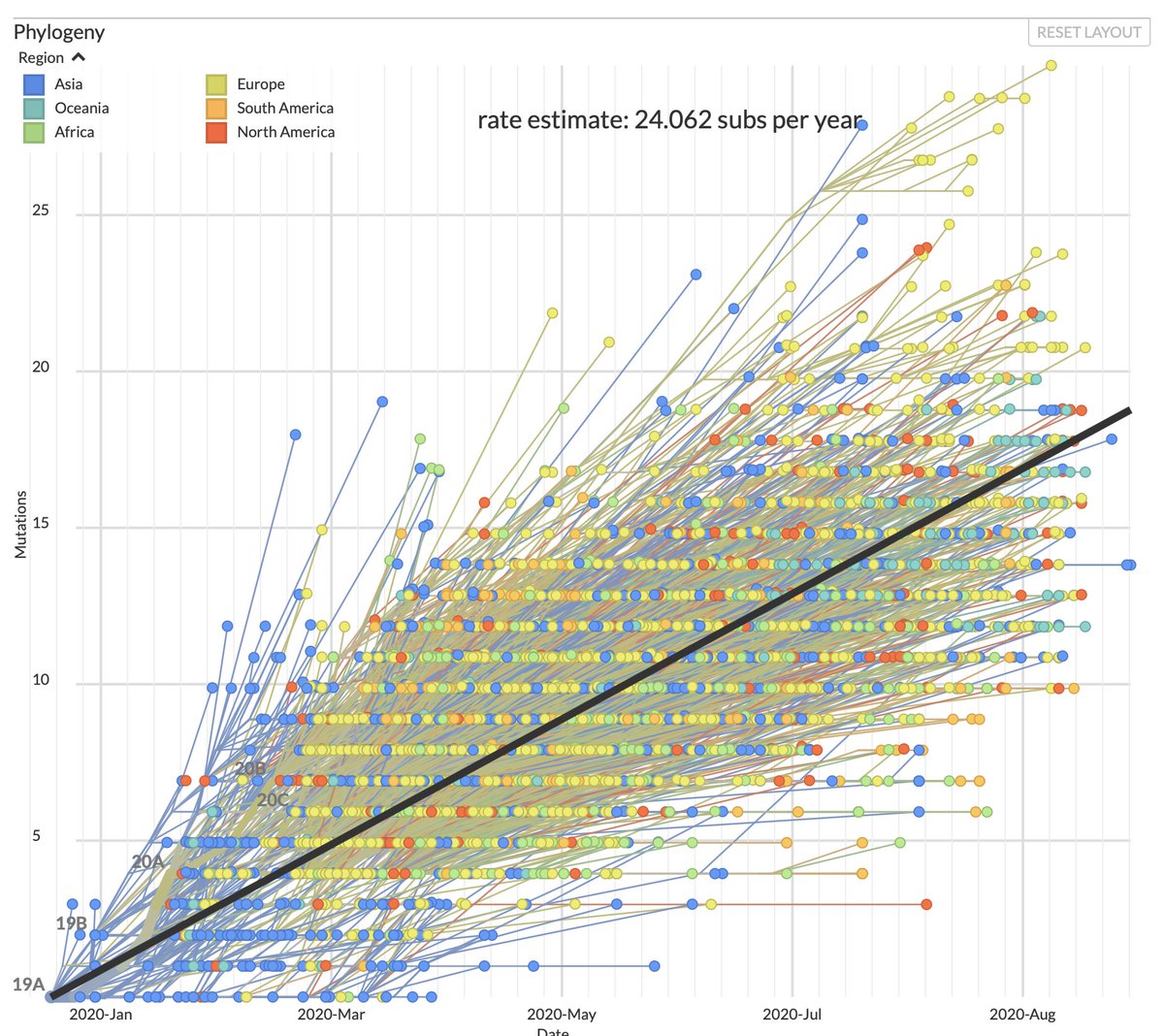
¿Debemos preocuparnos por estas mutaciones? Mutar es parte normal del ciclo de vida del virus y aún no tenemos evidencia clara que alguna de estas mutaciones generen cambios en la virulencia o transmisibilidad del virus. 23/ nature.com/articles/s4156…
¿Y la mutación D614G? Hay evidencia que la forma G (que ahora predomina en la mayoría de países) hace al virus más transmisible en el laboratorio. Pero no sabemos si esto se traduce en mayor infectividad en la vida real. El debate continúa. 24/ nytimes.com/2020/06/12/sci…
Vemos que la mayoría de genomas en Perú presenta la mutación G614 pero también hay variantes D circulando en junio. Vemos también que un aislado del INS parece haber mutado de vuelta a la forma D614, pero sin los datos crudos (.fastq) no podemos verificar esta observación. 25/ 

Por último, vemos que Lima y Callao presentan la mayor diversidad genética del virus, y que variantes observadas en Lima aparecen días/semanas después en otras regiones. Esto confirma que Lima y Callao fueron el origen de la epidemia en Perú. 26/ 

Noten que solo tenemos información de 11 de 25 regiones. En las próximas semanas INS nos va a dar 200 muestras de regiones para poder mejorar nuestro muestreo. 27/ 

Además, tenemos varios laboratorios regionales que han empezado a desarrollar su propia capacidad de diagnóstico molecular. Aquí entra el componente de descentralización y entrenamiento del proyecto. 28/
https://twitter.com/INS_Peru/status/1311456014919753730
En Lima, se une el grupo del Dr. Lenin Maturrano de UNMSM, que ha ganado un fondo concursable que nos permitirá secuenciar 400 muestras adicionales. Estos tres laboratorios cuentan con secuenciadores Illumina y se convertirán en nuevos puntos de vigilancia de esta red. 29/
En noviembre, organizaremos un taller de 5 días en UPCH para compartir experiencias y protocolos de secuenciamiento + análisis bioinformático. En diciembre/enero viajaremos a Arequipa y Chachapoyas para asesorarles en el secuenciamiento de nuevos genomas en sus laboratorios. 30/
¿Qué mas podemos hacer con estos genomas? Varios países ya han empezado a usar esta información para monitorear segundas olas de casos y brotes post-cuarentena. nature.com/articles/d4158… 31/
Un claro ejemplo es Nueva Zelanda, donde el gobierno utilizó información genómica para controlar el reciente brote en Auckland. Esto, por supuesto, va de la mano de un sistema robusto de rastreo de contactos. Aspiremos a ser como NZ. 32/ [
https://twitter.com/ESRNewZealand/status/1296308546582142978]
En 2021 o 2022 tendremos una o varias vacunas disponibles en Perú. Desde el punto de vista evolutivo, esto constituye un experimento gigante de presión evolutiva sobre el genoma del virus. 33/
Debemos mantener la vigilancia en los siguientes meses y años para identificar variantes que puedan "escapar" de vacunas, fármacos o el sistema inmune. También para identificar formas más o menos virulentas o transmisibles que aparezcan en el tiempo. 34/ nature.com/articles/d4158…
Cuando todo esto acabe, esta red de laboratorios pueden convertirse en la base de un sistema de vigilancia nacional que permita identificar tempranamente al siguiente virus de potencial pandémico. Debemos estar mejor preparados para la siguiente. 35/ nature.com/articles/nrg.2…
Financiamiento: antes mencionamos que cada genoma nos cuesta unos S/600 en reactivos y consumibles. Esto sin contar equipos, mantenimiento, servidores y pago a los estudiantes del equipo. 36/
[
[
https://twitter.com/pablotsukayama/status/1283892016406044672]
Aparte de Fondecyt, tenemos fondos de UPCH, UNMSM, BID y VLIR, por casi S/1,000,000. Seguimos aplicando a todo grant que aparezca, pero ya estamos agotando las vías tradicionales de financiamiento. Tenemos presupuesto para mantener la red de vigilancia hasta marzo 2021. 37/
Creemos que este es el momento de tocar las puertas de empresas, sociedades, fundaciones y fondos privados que nos permitan continuar este trabajo hasta 2022. Por favor, ayúdennos a llegar a ellos! 38/
Eso es todo (por ahora)! Seguiremos informando! Gracias a todxs los que nos apoyan. Gracias especiales a este grupo de jóvenes científicxs (faltan varios en la foto) que han sacado adelante este proyecto en condiciones muy difíciles. Son el futuro y corresponde apoyarles. 39/ 

Ah si:
#SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo 🇵🇪
#SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo #SinCienciaNoHayFuturo 🇵🇪
Aquí el post del lanzamiento del proyecto en julio.
https://twitter.com/pablotsukayama/status/1283891994914361345
¡Gracias por el apoyo y por compartir! Esta mañana estuvimos en @tvperupe hablando sobre genomas y evolución 😎 #SinCienciaNoHayFuturo
https://twitter.com/noticias_tvperu/status/1314561634644492289
• • •
Missing some Tweet in this thread? You can try to
force a refresh


