
Sobre vigilancia genómica del coronavirus y la nueva variante del Reino Unido.
Resumen: La estamos buscando pero se acabaron los fondos. #SinCienciaNoHayFuturo🇵🇪
1/n
Resumen: La estamos buscando pero se acabaron los fondos. #SinCienciaNoHayFuturo🇵🇪
1/n

La vigilancia genómica es el análisis sistemático de genomas de patógenos para entender su evolución y transmisión a escala local e internacional. nytimes.com/es/interactive…
2/
2/
SARS-CoV-2 es, de lejos, el patógeno más estudiado en la historia. En 12 meses de pandemia se han generado casi 300 mil genomas en más de 100 países que han sido depositados en bases abiertas para que todos las puedan analizar. gisaid.org
3/
3/
UK, donde se origina esta nueva variante, creó en marzo un consorcio de laboratorios públicos y académicos con £20 millones para vigilancia genómica de SARS-2. Hoy han secuenciado 160,000 genomas, casi el 9% de todos sus casos de Covid-19. cogconsortium.uk @UKinPeru
4/
4/
Es gracias a este sofisticado sistema de vigilancia que han podido rastrear el origen y aumento explosivo de una nueva variante en las últimas semanas. virological.org/t/preliminary-…
5/
5/
1.Rápidamente ha reemplazado a otras variantes del virus en UK.
2.Presenta varias mutaciones en regiones importantes para el reconocimiento del virus.
3. Algunas de estas mutaciones ya han mostrado (en el laboratorio) mayor capacidad del virus de infectar células humanas.
7/
2.Presenta varias mutaciones en regiones importantes para el reconocimiento del virus.
3. Algunas de estas mutaciones ya han mostrado (en el laboratorio) mayor capacidad del virus de infectar células humanas.
7/
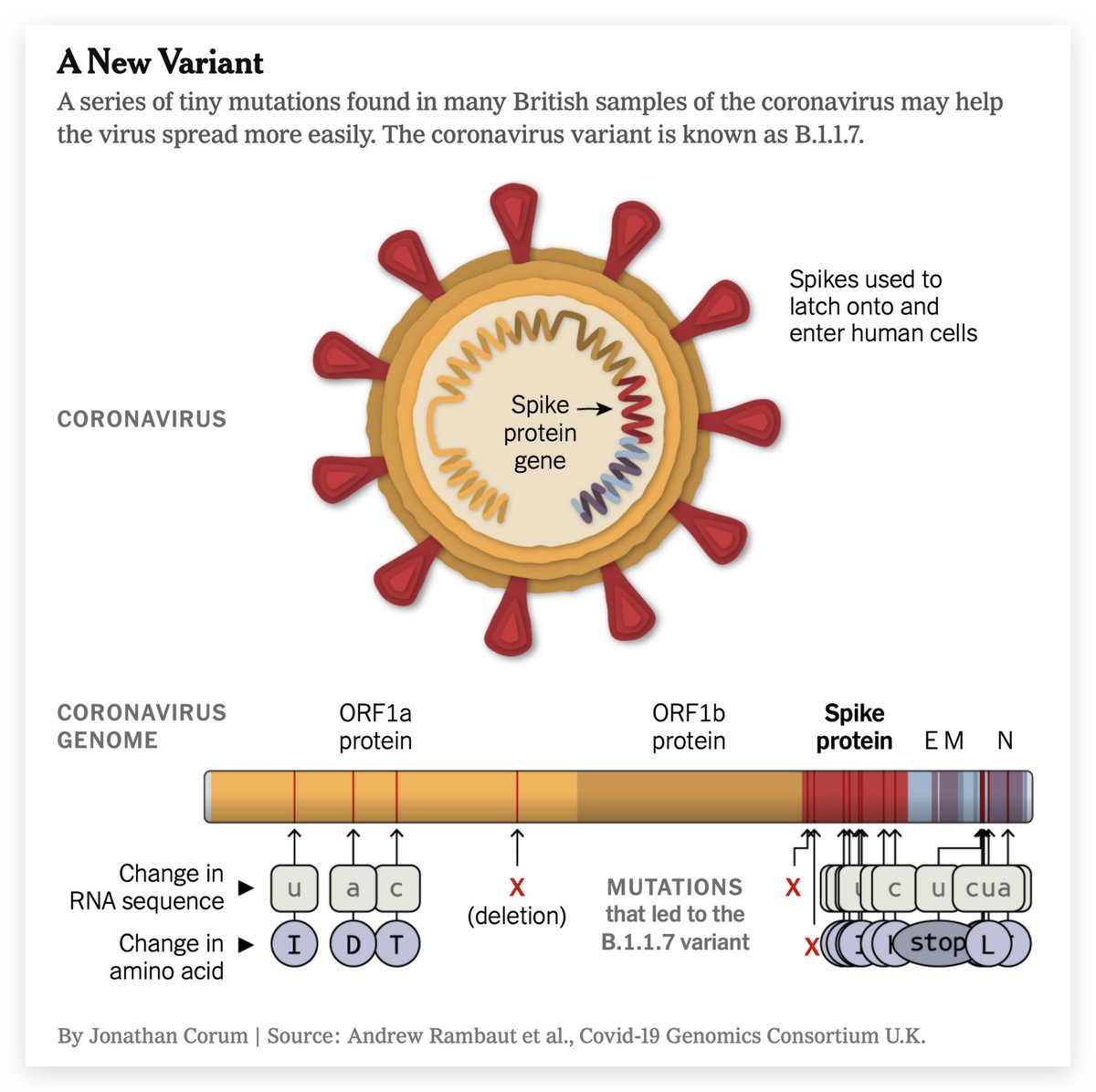
Aquí más detalles de esta nueva variante.
nytimes.com/2020/12/21/hea…
8/
https://twitter.com/mugecevik/status/1341094836682838021
nytimes.com/2020/12/21/hea…
8/
Mediante secuenciamiento de genomas, otros países ya han empezado a reportar casos con B.1.1.7. No sabemos cuál es la situación países que no tienen esta capacidad de vigilancia. cov-lineages.org/global_report.…
9/
9/

También se ha reportado una nueva variante dominante en Sudáfrica, diferente a la de UK, pero que comparte algunas de las mutaciones que preocupan. Se le denomina B.1.351.
medrxiv.org/content/10.110…
10/
medrxiv.org/content/10.110…
10/
UK y SA detectan nuevas variantes y suenan alarmas tempranas porque son los países que más hacen vigilancia en sus regiones. El resto de países, en la medida de sus posibilidades, deberían estar haciendo lo mismo. No lo digo yo, lo dice la OMS.
11/
https://twitter.com/Chjulian/status/1341172927455240193
11/
Hace 6 meses implementamos un sistema de vigilancia genómica de coronavirus en Perú, en colaboración con @INS_Peru y varias universidads peruanas.
12/
https://twitter.com/pablotsukayama/status/1283891994914361345
12/

Aquí aprovecho nuevamente para agradecer al increíble grupo de investigadorxs que ha hecho posible este trabajo en pandemia. Son lo máximo. @quipupe @Guillermo_S_S_R @davilabarclay @reymonera @janetrhm @PoolMARCOSCARB1 @TropicalesUPCH
13/

13/


En las últimas semanas hemos trabajado con colaboradores de @pucp @INS_Peru @untrm @UNSA_Oficial en la transferencia de protocolos de secuenciamiento, para que puedan ser replicados y analizados en sus laboratorios.
14/



14/




Esta semana subimos a @GISAID 58 nuevos genomas de muestras colectadas hasta octubre en Lima y Amazonas. Entre INS y UPCH tenemos hoy 355 genomas peruanos publicados, que corresponden al 0.03% del millón de casos oficiales de covid-19 en Perú.
15/



15/




Pueden explorar los datos en este build de @nextstrain (n=338) preparado por @quipupe nextstrain.org/community/quip…
16/
16/
Hasta octubre hemos detectado 26 linajes de SARS-CoV-2 circulando en Perú. No hemos encontrado las variantes B.1.1.7 y B.1.351 en nuestras muestras. Es poco probable que las variantes hayan ingresado y transmitido activamente en esas semanas. Necesitamos secuenciar más.
17/

17/


Aquí una comparación rápida con linajes circulando en Colombia.
18/
https://twitter.com/jdramirez24/status/1341503139527217162
18/
Aquí otra forma de visualizar los datos, via TreeToM. En unos días preparamos un hilo más detallado sobre los últimos hallazgos en estos genomas peruanos.
19/
19/

Esta semana nos reunimos con INS. El lunes nos darán muestras de 100 casos de Lima de diciembre. Pedirán muestras de viajeros positivos y de regiones que reportan aumento de casos. Tomará 2-3 semanas tener los resultados. Recién ahí la tendremos más clara.
20/
20/
Hay mucho que aún no se sabe sobre esta variante. Pero lo que se sabe, preocupa. Se necesitan experimentos en laboratorio y modelos animales para confirmar sus propiedades. Estudios están en marcha en todo el mundo pero tardarán semanas en publicarse.
21/
21/
Por mientras, aplican los mismos cuidados que para otras variantes del virus: evitar reuniones y lugares concurridos, ventilación, mascarilla, lavado de manos.
22/
https://twitter.com/trishgreenhalgh/status/1340677485856514050
22/
Esta no va a ser la última variante que preocupe al mundo. Mientras la gente se siga infectando el virus seguirá mutando. Las mismas condiciones que permiten su aparición en UK y SA están presentes en países con poblaciones grandes y alta transmisión, como Perú.
23/
23/
Pronto empezaremos a vacunar a buena parte de la humanidad. Hay que estar atentos a las nuevas variantes que se originen en todo el mundo en respuesta a las vacunas y a nuevos tratamientos que se introduzcan en 2021.
24/
24/
Necesitamos un sistema robusto de vigilancia genómica que nos ayude a manejar mejor esta pandemia y nos permita estar más preparados para la próxima.
25/
25/
Ahora, hablemos de fondos. El proyecto ha sido financiado por Fondecyt, la Cooperación Belga y fondos internos UPCH, por casi S/500,000. Con estos 100 últimos genomas ya nos quedamos sin materiales para seguir procesando muestras en 2021.
26/
26/
Necesitamos aprox S/2 millones para escalar el sistema de vigilancia y generar información hasta 2022. Postulamos a grants nacionales y extranjeros. También a Proyectos Integrales de Fondecyt, que financiaba escalamiento de proyectos Covid, pero nada.
fondecyt.gob.pe/convocatorias/…
27/
fondecyt.gob.pe/convocatorias/…
27/
Consideramos que esto ya dejó de ser un proyecto de investigación y se convierte en una herramienta de salud pública en la que vale la pena invertir. Porque #SinCienciaNoHayFuturo pero #SinFondosNoHayCiencia ✊
28/
28/
Por eso, un último intento: ¿Conocen de fundaciones, sociedades, empresas u otros fondos privados que nos puedan ayudar a mantener y escalar este sistema en los próximos meses? Toda ayuda es bienvenida. ¡Gracias!
FIN.
29/
FIN.
29/
Hace unos días en #RPPNoticias
Y un video sobre el proyecto que nos preparó @InvestigaUPCH para #PeruConCiencia
• • •
Missing some Tweet in this thread? You can try to
force a refresh





