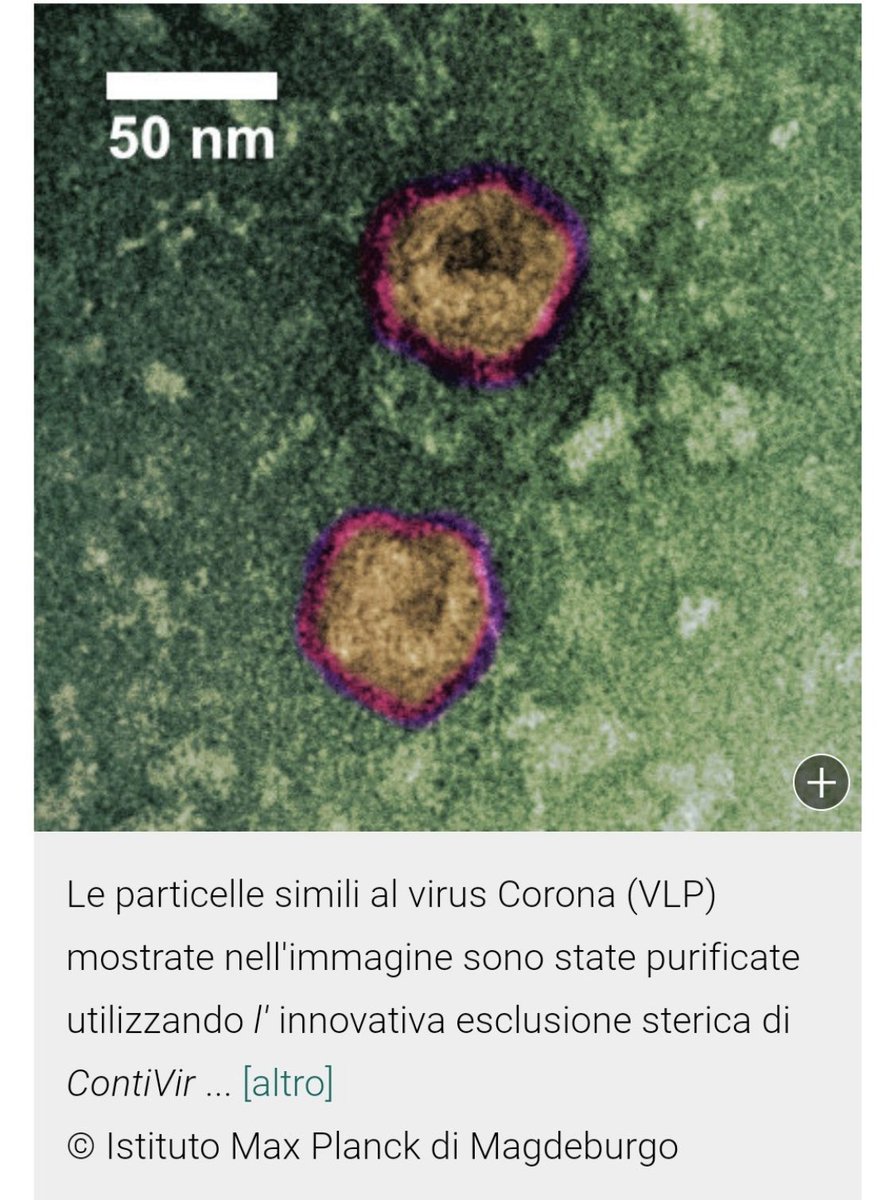
Hace muchos meses, intentando entender por qué el virus de la Pif estaba involucrado en SC2, mi línea de estudio no era muy ortodoxa. Creaba comparativas así. Mi forma de estudiarlo es rudimentaria. Ridícula para científicos (normal) pero gracias a ella, comencé a ver “trampas”👇 

Sabía que en febrero 2020 habían ocultado documentos pero no tenía sentido que en virus de humanos (SARS) se molestaran en ocultar o cambiar datos del virus del gato. Mis estudios sobre Pif, siempre se han centrado en su patogénesis, adentrarme en un genoma eran palabras mayores
Pero lo hice. Intentando aprender cómo estaba construido el genoma “origen” vi 4 aminoácidos que reconocí “rápido” en Pif.
Se habían estudiado mucho así que busqué los documentos que recordaba sobre ese motivo de secuencia S1/S2 en Pif. “Reinaba Trump” y no encontraba NADA🤷♀️
Se habían estudiado mucho así que busqué los documentos que recordaba sobre ese motivo de secuencia S1/S2 en Pif. “Reinaba Trump” y no encontraba NADA🤷♀️
Pero algo cambió (dejémoslo ahí) a inicios de 2021 y aparecieron documentos que daba por borrados. Los estudios en la Pif sobre esta proteína Spike eran tantos desde hace años que entre ellos ví este exhaustivo y revelador para mí, entre mis notas: wwwnc.cdc.gov/eid/article/19… 



Entonces traté de decir la coincidencia de esos AA con respecto a SC2 pero para los científicos el virus de gato solo era un Alphavirus con solo una coincidencia de 53% y no perdían tiempo con él.😔 No importaba que justo en esos AA encajara al 100% y documentado, daba igual 😳
Tampoco importaba que hubieran estudiado la Pif científicos vinculados a SARs/MERs/UNC/OMS/Wuhan y ahora SC2, solo era un 53% y punto. Como yo entendía que, por lógica, debía de haber alguna relación seguí estudiándolo. Empezar a entender “un genoma” era locura pero podía ayudar.
No voy a entrar en las dificultades ni irregularidades que vi al utilizar herramientas comparativas (Blast, Gisaid) ni las extrañas actualizaciones a 2020 de virus que no se actualizaban desde hacía años. Pero a los científicos les parecía normal y no había nada raro que pensar🧐
En este virus todo va rápido así que inicié un estudio sobre esos aminoácidos que saqué en este hilo pero ante el poco interés que despertaba, mis torpes formas de decirlo y mil cosas por estudiar sobre alimentos y prevención lo dejé apartado. Pero apareció Nicola y lo leyó ✨♥️
https://twitter.com/martinasisters/status/1359809283643342850
Como me sucedió con Dra Addie, él también creyó en mí, me dio fuerzas y avancé. Como bien dicen los trabajos de @BidoliNicola los científicos saben bien cómo hacer insertos, NSP16 cuadra en Pif y PRRAR encaja bien con otro patógeno. Tenía lógica, pero para mí faltaba algo más.
En este documento de Arxiv se explica la importancia de zona de corte y se estudia qué sucede cuando en esa zona se varían los aminoácidos. Esto es clave. Resulta ser zona S1/S2 en la que si se cambia algún AA en algunas posiciones se produce cambio en S2’ arxiv.org/pdf/1412.4034.… 

El serotipo I de Pif NO se propaga en cultivo celular. El serotipo 2 sí y fácilmente. Esto es importante porque el genoma que yo vi actualizado a 2020 fue el 79-1146, no el 79-1183. No entendía por qué un serotipo II ¡de laboratorio! sin coincidir en Spike con SC2, lo “tocaban”.
Ahora ya lo entiendo, su estructura S|P6-P1|S es exacta y “artificial”. Al conocer la variante India con mutación en P me ha llamado mucho la atención que esa mutación P681 sea por R (Arginina). UK ya me mosqueó con el P681H. He recordado estudio que hablaba de P6 en S1/S2 en Pif
En este estudio de Chang et alii, la posición P6 tiene importancia ya que se da un alto grado de conservación en ella en la posición de clivaje y eso resulta importante para la eficacia del corte por furina. ncbi.nlm.nih.gov/pmc/articles/P…
Se ha estudiado que P6 está muy bien conservado.
Se ha estudiado que P6 está muy bien conservado.
Según el documento de Arxiv, con 84 muestras de 39 gatos, se ha observado otra constante, un requisito mínimo para producir el corte por furina: debe haber una arginina (R) en posiciones P1 y P4, flanqueadas por residuos de Serina y residuo básico en P2.
P3 y P5 no son esenciales

P3 y P5 no son esenciales


Y el documento de Arxiv dice que debe haber escisión ¡por pares básicos! Los residuos básicos son: R,K,H.
Además los residuos intermedios deben constar de 0, 2 o 4 aminoácidos. zora.uzh.ch/id/eprint/1116…
Un consensus:
S|RXK/RR|S
(R es básico, X es algún residuo y K básico/lisina).
Además los residuos intermedios deben constar de 0, 2 o 4 aminoácidos. zora.uzh.ch/id/eprint/1116…
Un consensus:
S|RXK/RR|S
(R es básico, X es algún residuo y K básico/lisina).

Pongamos el FIPV (Pif):
SPRRARRS
S-Serina
P= P6
R= P5
R= P4
A= P3
R= P2
R= P1
S-Serina
Y el de SC2:
SPRRARS
Aguas abajo:
S
P= P6
R= P5
R= P4
A= P3
R= P2
❌= P1
S
Aguas arriba:
S
P= P6
❌= P5
R= P4
R= P3
A= P2
R = P1
S
¿Qué falla?
¡Exacto! ¡¡Faltaría UNO!🤔
SPRRARRS
S-Serina
P= P6
R= P5
R= P4
A= P3
R= P2
R= P1
S-Serina
Y el de SC2:
SPRRARS
Aguas abajo:
S
P= P6
R= P5
R= P4
A= P3
R= P2
❌= P1
S
Aguas arriba:
S
P= P6
❌= P5
R= P4
R= P3
A= P2
R = P1
S
¿Qué falla?
¡Exacto! ¡¡Faltaría UNO!🤔
Entonces revisé la documentación sobre SARs-CoV-2 para ver cómo tratan esa zona, cómo resuelven ese “desvío de consensus”.
Mirad OC43/HKU1 (2ª y 3ª línea)
S|SRRKRR|S
Arriba: Alinean secuencias y una R se sale
Abajo: no
Es un PREPRINT (bloqueado?) 🤔
researchgate.net/publication/33…
Mirad OC43/HKU1 (2ª y 3ª línea)
S|SRRKRR|S
Arriba: Alinean secuencias y una R se sale
Abajo: no
Es un PREPRINT (bloqueado?) 🤔
researchgate.net/publication/33…

Entonces miro más y, aunque lleva años documentado en felinos que los residuos P1 y P4 son los importantes, leo en otro documento que en SC2 “determinan” que P3 es residuo necesario😳, cuando en Pif ha demostrado ser residuo no crítico. ¿En qué quedamos? biorxiv.org/content/10.110… 



Al comparar en otros las secuencias veo con asombro que a pesar de que la estructura S|RXK/RR|S (consensus RRxRR) es altamente conservada en virus comparados, SC2 es “especial” ¡porque lo ajustan! no solo en HKU1 sino en OC43 y otros🧐 Buscad comparativas entre virus y lo veréis. 

Por tanto, SC2 sigue estudios de FIP de Arxiv y de Chang et al. (2012) AL PIE DE LA LETRA”🤔
Pero como SC2 tiene residuos R descompensados😔 para que les cuadre dicen que P3 es residuo de R necesario bajo el consensus SP/RxxS.
Y bloquean documentos con consensus RRxRR 🤔


Pero como SC2 tiene residuos R descompensados😔 para que les cuadre dicen que P3 es residuo de R necesario bajo el consensus SP/RxxS.
Y bloquean documentos con consensus RRxRR 🤔



Es decir, para que SC2 sea virus “especial” hallan un P3 “necesario” pero desprecian toda la documentación de años de la Pif. 😔
Y ahora, la 💣💥:
Encuentro un documento 2020 publicado revisado por pares, PERO con gráficas que NO tienen HKU1 ni OC43 😳
Claro ¡no cuadrarían! 😔

Y ahora, la 💣💥:
Encuentro un documento 2020 publicado revisado por pares, PERO con gráficas que NO tienen HKU1 ni OC43 😳
Claro ¡no cuadrarían! 😔


¡FALTA UNA R! (Arginina). Las R están descompensadas en el virus entregado por Wuhan (PRRAR) y mucho más descompensadas en la variante descubierta en India (RRRAR) en la que se ha perdido Prolina (P) y ha sido sustituida por residuo básico. ¡La R aumenta divisibilidad celular! 😔
Recordemos: requisito mínimo en FCS: debe haber básicos (R,K,H) en P1 y P4 y básico en P2.
P3 intermedio no esencial.
P6= P ahora es R en 🇮🇳‼️
P5= ?? 😳
P4= R
P3= R ¿necesario? 😳
P2= A
P1 = R ??
¡No cuadra, según su hipótesis falta P5, y según consensus falta P1! ¡Una R!

P3 intermedio no esencial.
P6= P ahora es R en 🇮🇳‼️
P5= ?? 😳
P4= R
P3= R ¿necesario? 😳
P2= A
P1 = R ??
¡No cuadra, según su hipótesis falta P5, y según consensus falta P1! ¡Una R!


Bien, pues aunque hay publicación de 2012 que fija esa estructura con un P3 no necesario, los científicos publican un “sorpresivo hallazgo” ÚNICO en SC2 de estructura entre Serinas con P3 necesario, en Nature, PERO... ¡solo ponen gráficas que cuadren! 😳😳 nature.com/articles/s4159…
NECESITAN descuadrar ese motivo de secuencia RRAR y dejarlo en 4 AA (no 5). Así SC2 es “único”. Pero con literatura científica de 2012, se puede ver que ¡son 5!, falta uno. ¡FALTA UNA R!
¿Escape de vacuna inacabada?
¿Con PRRARR es Pif?
¿Con RRARR deja de infectar a humanos?‼️🙏
¿Escape de vacuna inacabada?
¿Con PRRARR es Pif?
¿Con RRARR deja de infectar a humanos?‼️🙏

@threadreaderapp compile
Unroll please 🙏
Unroll please 🙏
• • •
Missing some Tweet in this thread? You can try to
force a refresh