En 🇦🇺 han publicado como sería su sistema de vigilancia genómica ideal para la #COVID19.
Un sistema global basado en #OneHealth, integrando la situación epidemiológica, las características de los brotes y las dificultades q conlleva la secuenciación.
Te cuento
#abroHilo 🧵
1/n
Un sistema global basado en #OneHealth, integrando la situación epidemiológica, las características de los brotes y las dificultades q conlleva la secuenciación.
Te cuento
#abroHilo 🧵
1/n

Australia ha sido uno de los países que mantuvo la estrategia zeroCovid (hasta omicron), secuenciando un alto porcentaje de las muestras +.
Y les ha permitido inferir transmisión local en circunstancias particulares. Por ejemplo en los hoteles cuarentena.
theage.com.au/national/victo…
Y les ha permitido inferir transmisión local en circunstancias particulares. Por ejemplo en los hoteles cuarentena.
theage.com.au/national/victo…
🔘Por el elevadísimo número de casos y laas altas tases de transmisión
🔘Por los cuellos de botella en el procesado bioinfomático y estadístico de los modelos
🔘Porque la vigilancia genómica por si sola no es suficiente para producir resultados que tengan sentido epidemiológico.
🔘Por los cuellos de botella en el procesado bioinfomático y estadístico de los modelos
🔘Porque la vigilancia genómica por si sola no es suficiente para producir resultados que tengan sentido epidemiológico.
La cantidad de genomas existentes de #SARSCoV2 en las bases de datos son unos 5 órdenes de magnitud superior a la de cualquier otro patógeno en la historia de la secuenciación. Esto ha provocado que la biocomputación no sea capaz de procesar tal cantidad de información.
Además, el formato en el que se guardan estas secuencias y sus metadatos ofrece un gran margen de mejora.
Y las bases de datos como GISAID tienen un importante sesgo de muestreo hacia las regiones donde más se secuencia: UK, Europa y EEUU.
Y las bases de datos como GISAID tienen un importante sesgo de muestreo hacia las regiones donde más se secuencia: UK, Europa y EEUU.
Tampoco hay directrices claras sobre como minimizar el impacto del sesgo de muestreo.
Así que han decidido desarrollar una estrategia que equilibre una secuenciación "representativa" con una "focalizada" para cubrir lo más rápido posible las nuevas VOC que puedan surgir.
Así que han decidido desarrollar una estrategia que equilibre una secuenciación "representativa" con una "focalizada" para cubrir lo más rápido posible las nuevas VOC que puedan surgir.
Esta secuenciación equilibrada considera:
⏹️Casos enfocados a brotes, viajeros, residencias, hospitalizaciones, y muertes.
⏹️Casos representativos: muestreo aleatorio de casos positivos.
⏹️Vigilancia en aguas residuales.
⏹️Vigilancia en animales salvajes y domésticos.
#OneHealth
⏹️Casos enfocados a brotes, viajeros, residencias, hospitalizaciones, y muertes.
⏹️Casos representativos: muestreo aleatorio de casos positivos.
⏹️Vigilancia en aguas residuales.
⏹️Vigilancia en animales salvajes y domésticos.
#OneHealth
En circunstancia normales, la secuenciación del 10% de los casos sería suficiente para informar en materia de salud pública.
En épocas de alta prevalencia en la población, un 1-2% de los casos podría ser suficiente.
En épocas de alta prevalencia en la población, un 1-2% de los casos podría ser suficiente.
La parte representativa (no-estratégica), debe enfocarse principalmente en un muestreo aleatorio de los casos (~90%). Un 10-15 % en viajeros, y otro 5-10% en vigilancia de aguas residuales. 
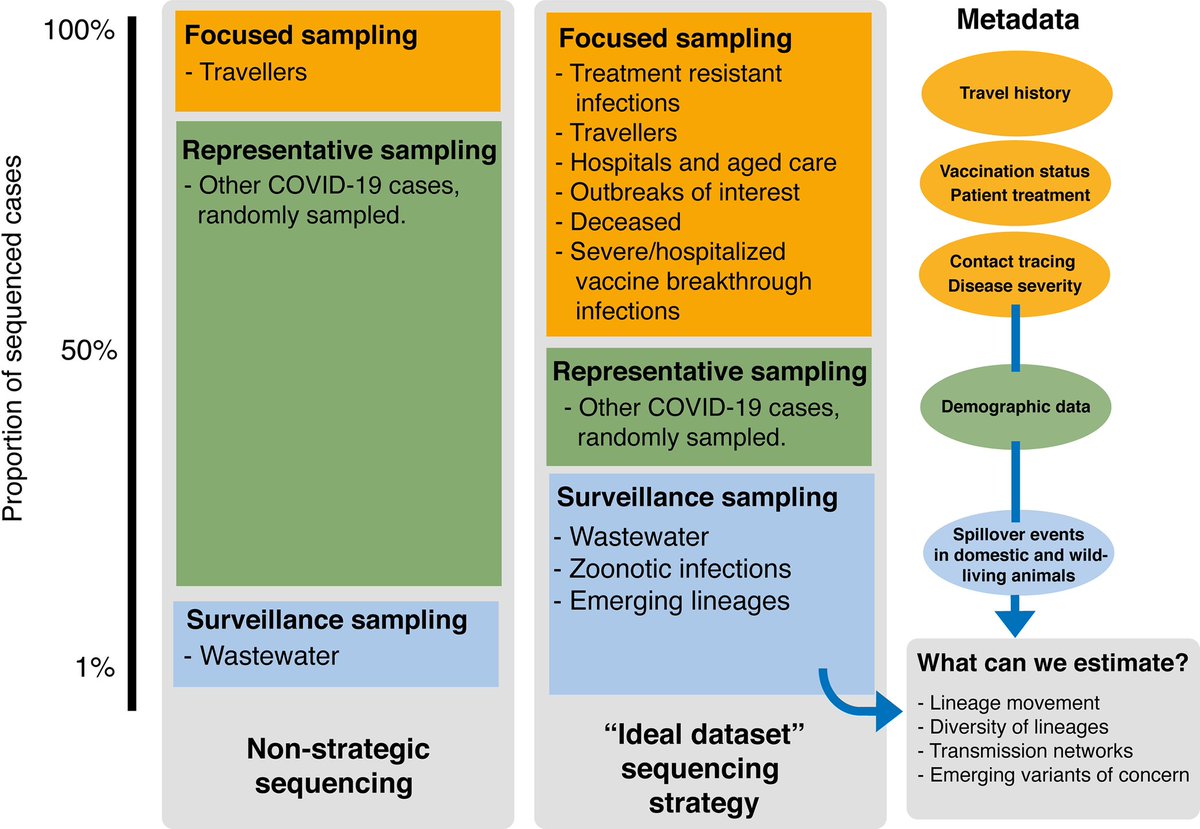
La parte estratégica debe ir dirigida en un 50% a hospitalizaciones, casos difíciles de tratar, decesos, hospitales, y residencias.
15% a otros casos aleatorios.
35% vigilancia en aguas residuales, animales y variantes emergentes.
15% a otros casos aleatorios.
35% vigilancia en aguas residuales, animales y variantes emergentes.
La secuenciación se realizará por los respectivos centros de cada región. Pero todas las secuencias serán centralizadas en un sistema accesible denominado "AusTrakka".
Cada secuencia debe ir acompañada de unos
* metadatos * recogidos de forma sistemática.
Cada secuencia debe ir acompañada de unos
* metadatos * recogidos de forma sistemática.
Estos metadatos deben incluir por lo menos:
⏹️Datos demográficos
⏹️Severidad de la enfermedad
⏹️Reinfecciones
⏹️Estado vacunal
⏹️Información de rastreadores y contactos
⏹️Tratamiento
En caso de viajeros
⏹️Historia de viaje
En caso de animales
⏹️Información zootécnica
⏹️Datos demográficos
⏹️Severidad de la enfermedad
⏹️Reinfecciones
⏹️Estado vacunal
⏹️Información de rastreadores y contactos
⏹️Tratamiento
En caso de viajeros
⏹️Historia de viaje
En caso de animales
⏹️Información zootécnica
Esta estrategia evitará una sobre-representación de casos hospitalizados, y mejorará la detección en la introducción de nuevas variantes, y la identificación de eventos críticos.
🟦Los muestreos aleatorios deben equilibrar las zonas geográficas y las diferentes densidades de población.
🟦Los equipos deben ser multidisciplinares (salud pública, clínicos, microbiólogos, biología molecular, biocomputación, y análisis genético).
🟦Los equipos deben ser multidisciplinares (salud pública, clínicos, microbiólogos, biología molecular, biocomputación, y análisis genético).
🟦Además, la secuenciación debe ser continua, en lugar de racheada.
🟦Y se deben usar las infraestructuras y conocimiento existente, y las creadas durante la pandemia, sin necesidad de mucha mayor inversión.
🟦Y se deben usar las infraestructuras y conocimiento existente, y las creadas durante la pandemia, sin necesidad de mucha mayor inversión.
¿Es esta la mejor estrategia? Puede que no para todo el mundo, pero al menos en Australia han hecho la reflexión de como lo van a plantear, y lo han plasmado en un artículo revisado por pares.
Este artículo esta disponible en 👇:
doi.org/10.1371/journa…
(fin)
Este artículo esta disponible en 👇:
doi.org/10.1371/journa…
(fin)
• • •
Missing some Tweet in this thread? You can try to
force a refresh