Pronto saldrán noticias sobre el estudio de ganancia de función (GoF) en #SARSCoV2 de la Boston University.
No voy a entrar en la parte ética y de permisos del estudio, pero voy a comentar la parte técnica y por qué quizás no habría que considerarlo GoF.
#abroHilo 🧵
1/14
No voy a entrar en la parte ética y de permisos del estudio, pero voy a comentar la parte técnica y por qué quizás no habría que considerarlo GoF.
#abroHilo 🧵
1/14
Lo que hacen los autores es introducir el gen de la proteína Spike de omicron BA.1 en el genoma de la variante original (denominada WT, de Wild Type) de Wuhan .
El resultados es producto de la ingeniería genética y sí es un virus creado en laboratorio. Lo han llamado OMI-S.
2/
El resultados es producto de la ingeniería genética y sí es un virus creado en laboratorio. Lo han llamado OMI-S.
2/
También usan la variante Omicron BA.1 (con su genoma completo) en la comparación. Así que una representación esquemática del genoma de los tres tipos de #SARSCoV2 probados sería esta:
3/
3/

Para llamarlo GoF, yo esperaría que el virus ganase alguna propiedad o función que no tenía antes. En este caso lo que evalúan es evasión inmunitaria y progresión de la enfermedad.
4/
4/
En los resultados, no se ve está ganancia de función. Mas bien es una pérdida de función de OMI-S con respecto a WT.
Infecta similar que WT los receptores ACE2, ...
5/
Infecta similar que WT los receptores ACE2, ...
5/
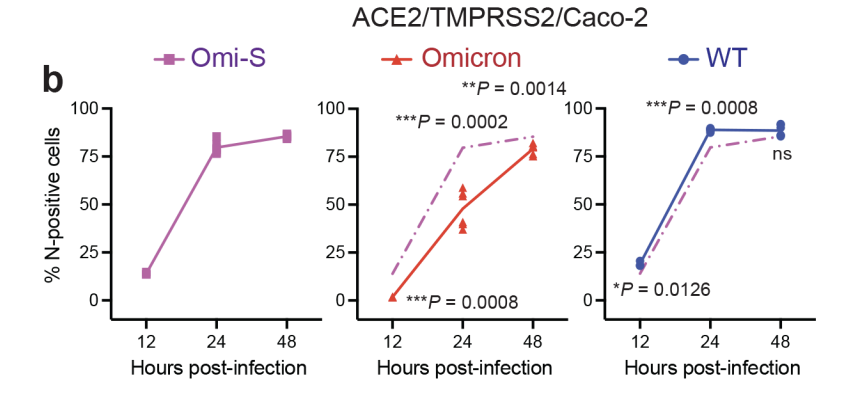
Omi-S tiene mayor capacidad infectiva que omicron.
Sí. Pero porque WT también la tiene.
Recordemos que la edición genética se hace sobre WT, y se le pone S de omicron. O sea que Omi-S "pierde" con respecto a su "backbone" que es WT.
8/
Sí. Pero porque WT también la tiene.
Recordemos que la edición genética se hace sobre WT, y se le pone S de omicron. O sea que Omi-S "pierde" con respecto a su "backbone" que es WT.
8/
Otro aspecto importante es q se usó una linea genética de ratón muy susceptible a #SARCoV2 (K18-126 hACE2).
Todos los 🐁🐁 infectados con WT murieron.
Mientras, todos los ratones infectados con Omicron sobrevivieron.
¿Qué pasó con los ratones infectados con el virus editado?
9/
Todos los 🐁🐁 infectados con WT murieron.
Mientras, todos los ratones infectados con Omicron sobrevivieron.
¿Qué pasó con los ratones infectados con el virus editado?
9/
que murieron el 80% de ellos. Es decir, sobrevivieron más que los infectados con la WT. Así que el virus editado tampoco ganó en mortalidad de estos ratones muy susceptibles a la enfermedad.
10/
10/

Por todo esto, mi *opinion* es que es un estudio con un virus editado y creado en laboratorio, pero no un estudio de ganancia de función.
Gente como @Virusemergentes, con quien he comentado el estudio, puede tener opiniones mucho más competentes que la mía.
11/
Gente como @Virusemergentes, con quien he comentado el estudio, puede tener opiniones mucho más competentes que la mía.
11/
Las 2 conclusión más importante que yo sacaría del estudio:
⏹️La variante WT es más peligrosa para los ratones del estudio que Omicron.
⏹️La patogenicidad del #SARSCoV2, va mucho más allá de la proteína SPIKE. (no todo es Spike)
12/
⏹️La variante WT es más peligrosa para los ratones del estudio que Omicron.
⏹️La patogenicidad del #SARSCoV2, va mucho más allá de la proteína SPIKE. (no todo es Spike)
12/
Como digo es mi opinión propia, y son los organismos competentes los que determinan si lo son o no.
De hecho el estudio ha sido aprobado por los comités de bioseguridad compentes.
13/
De hecho el estudio ha sido aprobado por los comités de bioseguridad compentes.
13/

Espero que os ayude a evaluar mejor las noticias que salgan en los próximos días.
Si te ha resultado interesante agradezco RT.
fin.
14/14
Si te ha resultado interesante agradezco RT.
fin.
14/14
• • •
Missing some Tweet in this thread? You can try to
force a refresh