@dr_l_alexandre
@TroncheBiais
Comme expliqué dans la vidéo ces affirmations reposent sur un article scientifique (préprint) d’une équipe indienne, déposé sur la plateforme BioRxiv (plateforme où l'on peut déposer des articles scientifiques avant qu’ils aient pu être révisés par des pairs).
L’objectif du papier consiste à démontrer que ce qui sépare génétiquement le SARS_COV2 de son cousin le plus proche, le SARS_COV1 de 2002, est l’existence de 4 petits fragments dans le gène de la protéine S. Et que c’est 4 fragments proviendrait du VIH.
Les auteurs déclarent ensuite que ces 4 fragments étaient aussi absents chez les coronavirus de chauve-souris.
Ce qui est pour eux « surprenant » car le SARS_COV2 était supposé provenir d’un coronavirus sauvage de chauve-souris.
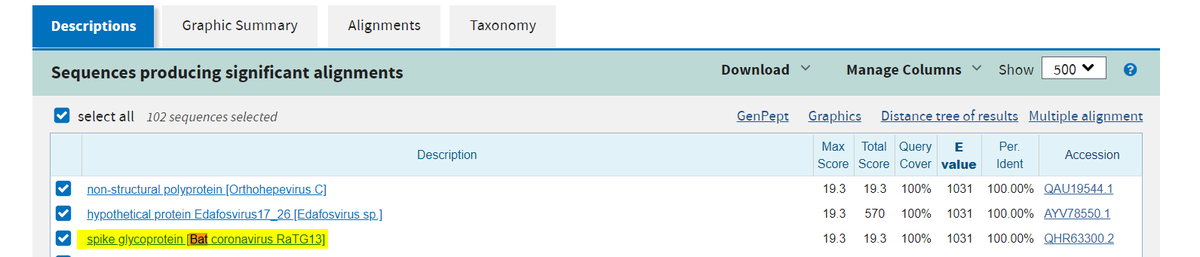
Pour enquêter sur l’origine de ces 4 fragments unique au SARS_COV2, les auteurs décidèrent d’utiliser un outil bio-informatique appelé BLASTp.
Grosso modo, cet outil permet de « scanner » le génome/protéome d’un grand nombre d’espèces pour en sélectionner des régions qui ont une forte homologie avec une séquence nucléotidique/protéique d’intérêt (ici les 4 fragments protéique).
C’est donc ce papier qui a fait dire à Luc Montagnier, prix nobel de médecine pour la découverte du VIH, que ce SARS_COV2 est probablement un virus modifié par l’œuvre de biologiste moléculaire…
C’est assez étonnant que l’info ne ressorte que maintenant via le passage TV de Montagnier car le papier date du 31 janvier.
Entre temps, chose rare, le préprint a été rétracté par les administrateurs de BioRxiv suite aux nombreux commentaires d’utilisateurs qui avait souligné (à juste titre) la faiblesse des affirmations faites par les auteurs.
Maintenant allons voir un peu si les résultats sont vraiment fiables !
Si on refait les expériences de BLASTp sur les 4 fragments on remarque que les auteurs se sont bien arrangés avec les résultats...
Les auteurs déclarent que les fragments proviennent du VIH?
Moi je déclare que les fragments proviennent de coronavirus de chauve-souris.
Et aux vues des résultats ces deux affirmations ont autant de force.
En fait cet méthode (le BLAST) n’est pas du tout adapté quand on utilise de si petite séquence sur de si grande base de données (ici tout le taxon virus).
Le % de tomber sur des séquences homologue par hasard est beaucoup trop élevé.
De plus dans la base de donnée utilisée (NCBI) on retrouve environ 6 millions de séquences de VIH contre 50k pour tout le taxon Coronaviridae (parce que le VIH est l’un des virus les plus étudié) ce qui augmente les chances de tomber sur une homologie avec le VIH.
Pour preuve si je Blast une séquence de même taille que celles du papier (7-8 acides aminés) prise au hasard je vais probablement observer des homologies avec le VIH.
En gros si on avait voulu confirmer l’idée préconçue que le coronavirus provient d’une manipulation humaine avec adjonction de séquence spécifique du VIH, on ne l’aurait pas mieux confirmer qu’en utilisant cette méthode…
En fait cette méthode est tellement pas adapté a la question posée par les auteurs que si je introduis un si petit fragment issue d’une espèce X dans la séquence d’une protéine d’un virus Y ...
... Vous ne serez jamais capable de retrouver l’espèce X en blastant le petit fragment contre l’ensemble des séquences connues protéiques du monde vivant.
C’est un peu comme si j’introduisais un mot d’un livre X dans un autre livre Y et que je vous demandiez de retrouver le livre d’où vient le mot introduit à partir d’une recherche sur google books…
C’est donc ce papier qui a fait dire à Luc Montagnier, prix nobel de médecine pour la découverte du VIH, que ce SARS_COV2 est probablement "un virus modifié par l’œuvre de biologiste moléculaire"…
...
Au cas où le smiley facepalm ou la suite du thread n'était pas assez clair :
C'est archi carrément complètement du hasard...
Ici, un fil twitter datant du 1er fevrier qui décrit plus formellement que parmi les 4 fragments "découvert" par les auteurs du préprint, 2 sont des artefacts et les 2 autres sont bien partagés avec des coronavirus de chauve-souris