Donc de la page 4 a 14 des supplementary information (sa fais toujours plaisir d'être reléguer en annexe des papier majeurs :) )
Y a des choses très intéressante :
Avec les reads de troisièmes génération on a un gros problème c'est le taux d'erreur important.
Le séquenceur ne vas pas lire les bonnes bases mais y a plusieurs type d'erreur différents.
Mais aussi des erreurs structurel:
- des "sequencing jump" on vas sauté une partie de la séquence.
- des chimères, on vas fusionné deux reads, donc relié deux régions du gènome qui n'ont aucun liens
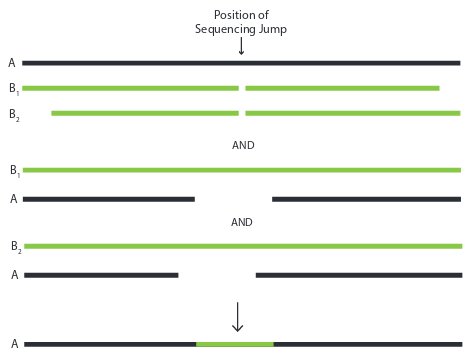
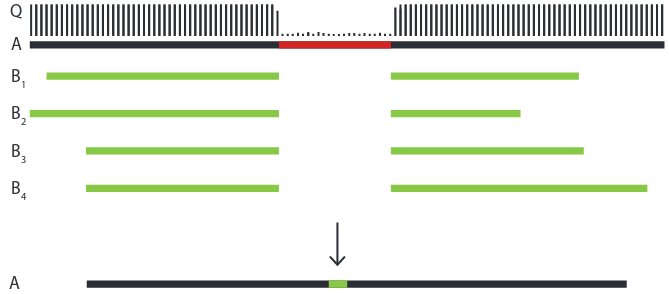
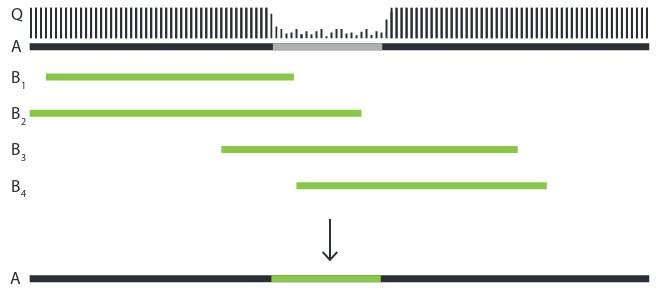
Les régions répété vue que ce sont des duplications on vas avoir beaucoup