
Comme promis, voici le thread sur les salmonelles ! Ici, je vais décrire rapidement les salmonelles et les classifications des salmonelles 😉
Alors la Salmonella, c'est quoi ? 🦠
Alors la Salmonella, c'est quoi ? 🦠
Entérobactérie ✅ Bacilles avec des flagelles péritriches (tout autour de la cellule) ✅Gram négatif ✅ 0,7 à 1,5 μm de diamètre✅Superbe photo coloré d'une Salmonelle en contact avec une cellule✅ (crédit NIAID) 

Elle provoque des maladies, comme la fièvre typhoïde chez les salmonelle typhiques, ou la salmonellose chez les salmonelles non-typhiques.
Dans mon étude, je me concentre sur les salmonelles non-typhiques, qui provoque des diarrhées, de la fièvre, et de grosses complications chez les personnes fragiles (notamment lorsque la salmonelle se retrouve dans le sang).
Et au niveau de la classification, ça donne quoi ?
Et au niveau de la classification, ça donne quoi ?
Salmonella est une très grande famille de bactérie. Elle contient 2 espèces, S.Bongori et S.enterica. La principale est S.enterica, qui est elle-même divisé en 6 sous-espèces. Je vous mets un schéma parce que ça devient compliqué. crédit : doi: 10.3389/fimmu.2014.00481 

Dans ces 6 sous-espèces, la plus connue est enterica, donc ça donne Salmonella enterica subsp. enterica. Jusque-là vous arrivez à suivre ? OK.
Là il faut imaginer qu'il y a encore un niveau dans la classification, qu'on appelle "sérovar".
Là il faut imaginer qu'il y a encore un niveau dans la classification, qu'on appelle "sérovar".
Un sérovar c'est quoi ? C'est une classification basé sur l'expression des antigénique de surface. C'est définis sur la base des antigènes somatiques O (lipopolysaccharide) et flagellaires H.
Pour simplifier, les salmonelles ont différentes combinaisons de type cellulaire et de flagelles comme illustré ci-dessous. Crédit : thevetgroup 

Typiquement, Salmonella présente deux phases flagellaires H, donc quand on décrit un sérovar de Salmonella ça donne "Salmonella (species) serotype (O antigen) : (H1 antigen) : (H2 antigen)".
Exemple : Salmonella enterica subsp enterica 1,4,[5],12:i:1,2. Si certains serovars sont connus, ils ont le droit à un nom. Par exemple, 1,4,[5],12:i:1,2 = Typhimurium. Et comme on est flemmard, on a le droit de dire S.Typhimurium pour aller plus vite.
Tout cela est décrit dans la bible des microbiologistes spécialisés en Salmonella : "Kauffman–White classification" de Pasteur de 2007. Voilà à quoi ça ressemble. 
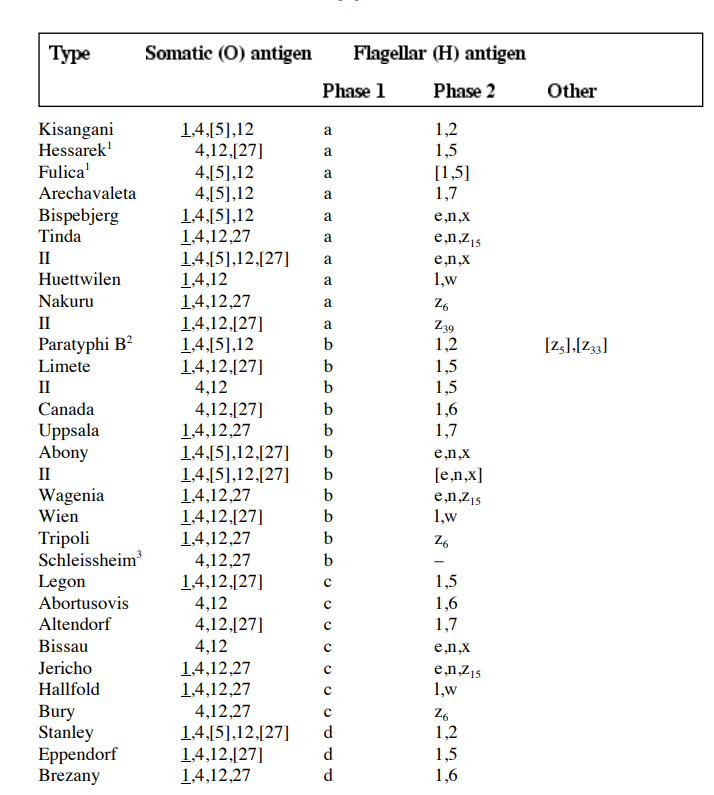
Mais pourquoi je vous parle de tout ça ?
Parce qu'en fait, des sérovars, il y en a 2600. C'est ENORME. Et ces sérovars, ils ont des hôtes différents et des profiles de virulences complètement différents.
Parce qu'en fait, des sérovars, il y en a 2600. C'est ENORME. Et ces sérovars, ils ont des hôtes différents et des profiles de virulences complètement différents.
Par exemple, S.Typhi est une Salmonella typhique qui se retrouve chez les humains, alors que S.Typhimurium est non typhique, et se retrouve principalement chez la volaille, le porc et l'humain.
Allez, je rajoute un petit coup et après promis, j'arrête.
Je vous ais dit qu'il y avait 2 phases flagelles (le H1 et le H2). Eh bien, chez S.Typhimurium, il arrive que la 2eme phase flagellaire ne soit pas exprimée. On a donc un variant monophasique de S.Typhimurium.
Je vous ais dit qu'il y avait 2 phases flagelles (le H1 et le H2). Eh bien, chez S.Typhimurium, il arrive que la 2eme phase flagellaire ne soit pas exprimée. On a donc un variant monophasique de S.Typhimurium.
Donc si S.Typhimurium est : 1,4,[5],12:i:1,2 , son variant monophasique est : 1,4,[5],12:i:-.
Si j'en parle, c'est parce que le variant monophasique de S.Typhimurium a été décrit pour la première fois dans les années 90, et depuis il est encore plus prévalent que S.Typhimurium. Pourquoi ? On ne sait pas encore.
Moi, je travaille principalement sur S.Typhimurium (donc Salmonella enterica subsp Typhimurium), son variant monophasique, et S. Mbandaka.
S.Typhimurium et son variant monophasique (1,4,[5],12:i:-) fait parti des sérovars les plus détectés en France et corresponds à 60 % des serovars de Salmonella retrouvés dans le porc.
Crédit : CNR Pasteur rapport de 2019.
Crédit : CNR Pasteur rapport de 2019.

D'ailleurs, S.Typhimurium et son variant monophasique font partit des sérovars hautement surveillé et déclaré "Sérovars d’intérêt en santé publique".
S.Mbandaka se retrouve ce sérovar chez les bovins, la volaille et l'humain. La transmission chez l'homme est plus rare que S.Typhimurium, mais elle a été responsable de plusieurs crises sanitaires, comme la contamination de céréales dans plusieurs pays en 2018.
Je vais m'arrêter ici pour la description des salmonelles ! Demain, je parlerais de la caractérisation et des enquêtes épidémiologiques des salmonelles lors d'une crise sanitaire ! Bonne soirée à vous
• • •
Missing some Tweet in this thread? You can try to
force a refresh



