En 2015, investigadores estadounidenses analizaron las muestras de heces de personas americanas, francesas, gambianas y hasta de simios del zoo de San Francisco. Todas las muestras
Al determinar el #virioma en la muestras de heces (todos los virus presentes), notaron que una alta incidencia de un virus desconocido para la ciencia.
Para que tengan una idea, el genoma del VIH es de 9.2 Kb, el de zika de 11 Kb y el del ébola de 18 Kb, aproximadamente.
Sin embargo, no pudieron infectar ratones con #smacovirus.
El paper es este: ncbi.nlm.nih.gov/pubmed/27774288
El profesor Mojica no lo hizo solo, como es habitual en la ciencia. Una persona clave fue
Ayer, César compartió en su Facebook la publicación de su más reciente paper en Nature, y tiene que ver con los smacovirus y CRISPR. Así que volvemos a la ciencia.
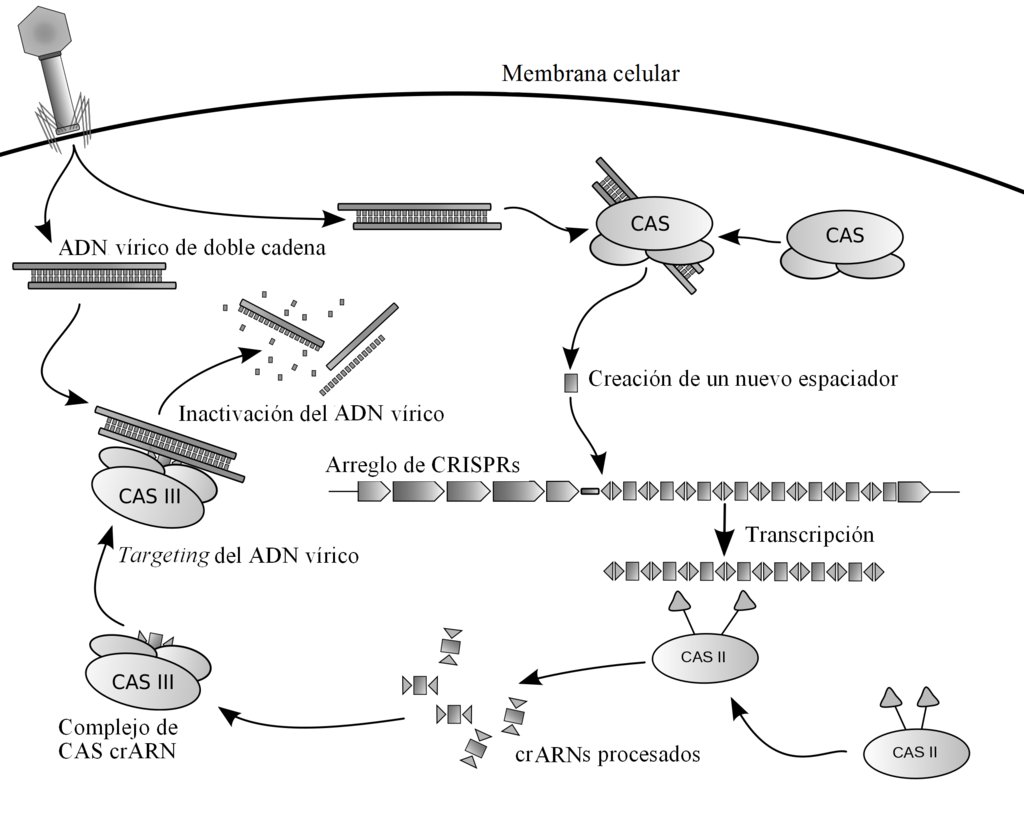
Conocer la secuencia de los espaciadores de las CRISPR hace posible asignar el par virus-huésped en procariotas de una forma bastante certera.
Con esto en mente, César hizo una búsqueda exhaustiva de genomas virales en espaciadores de CRISPR.
Encontró, casi por serendipia, una pareja perfecta entre los espaciadores de CRISPR de una arquea metanogénica y un smacovirus.
La arquea es Candidatus Methanomassiliicoccus intestinalis, un habitante de nuestros intestinos.
Y sugiere que, de alguna forma aún desconocida, esta infección entre microorganismos en nuestros intestinos puede ocasionar diarrea.
Lo que hace maravilloso este paper, entre otras implicaciones, es que confirma nuestros puntos ciegos sobre la vida microbiana, su diversidad y su ecología.
Yo alucino y felicito a César Díez-Villaseñor, codescubridor de las CRISPR, por este aporte.
El paper es este: nature.com/articles/s4146…
#ComunicaCiencia #Microbiología
El propio César ha hecho una reseña en @NatureMicrobiol sobre su trabajo.
naturemicrobiologycommunity.nature.com/channels/346-b…