
Sobre vigilancia genómica y variantes importadas vs. variantes locales del virus. 🧵
1/13
1/13
El secuenciamiento genómico nos sirve para detectar el ingreso de las nuevas variantes B.1.1.7 🇬🇧, B.1.351 🇿🇦 y P.1 🇧🇷, pero también para monitorear la transmisión y evolución de las *cientos* de variantes que ya circulan activamente en el país desde hace meses.
2/13
2/13
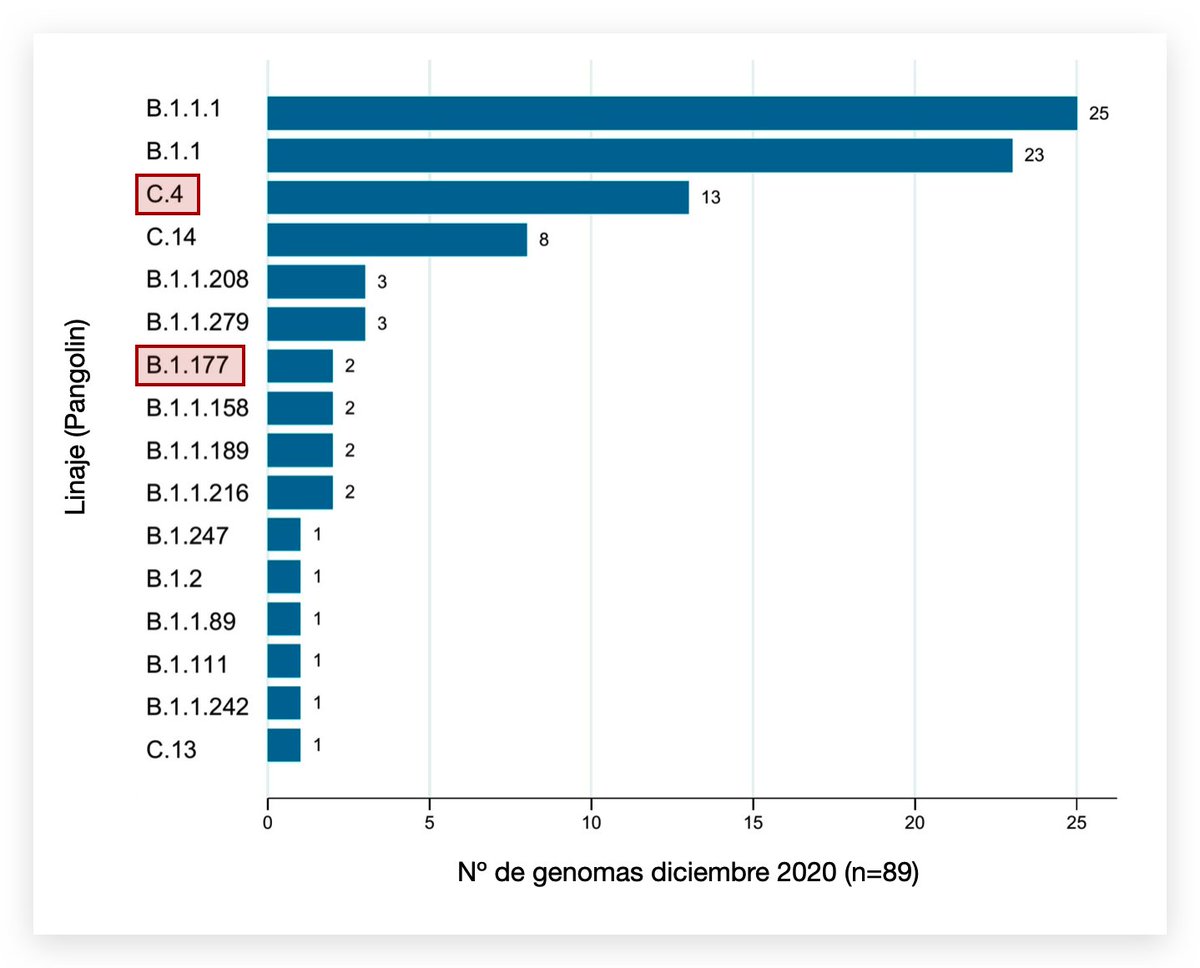
La semana pasada compartimos los resultados de nuestro último muestreo. No encontramos dichas variantes pero detectamos 16 linajes circulando en Lima en diciembre.
Hoy les voy contar el sobre B.1.177 🇪🇸 y C.4 🇵🇪
3/13

Hoy les voy contar el sobre B.1.177 🇪🇸 y C.4 🇵🇪
3/13
https://twitter.com/pablotsukayama/status/1350423096583462914
B.1.177 (también llamada 20A.EU1) es una variante que apareció en España alrededor de junio y se transmitió rápidamente por todo Europa gracias a la reapertura de vuelos en la UE y los veraneantes que viajaron a España.
4/13
4/13
https://twitter.com/firefoxx66/status/1321725353371586560
Se caracteriza por la mutación A222V en el dominio NTD de la proteína Spike y hoy es la variante predominante en varios países de Europa. Todavía se discute si es más transmisible o no.
5/13
medrxiv.org/content/10.110…

5/13
medrxiv.org/content/10.110…


Pues, resulta que desde octubre hemos detectado en Lima 3 genomas B.1.177. Esto sugiere que la variante ha ingresado en múltiples ocasiones luego de la reapertura de vuelos internacionales. Hoy se transmite activamente en Lima. Puede ocurrir lo mismo con la variante #B117🇬🇧.
6/13

6/13

Ahora miremos hacia adentro y hablemos de la variante C.4. Esta la vemos en Perú desde junio. En diciembre fue el tercer linaje más abundante en Lima, con 13/89 de muestras (12%). Fuera de Perú, solo ha sido reportada una vez en Serbia, en junio.
7/13
cov-lineages.org/lineages/linea…
7/13
cov-lineages.org/lineages/linea…

C.4 se caracteriza por la mutación A1078S en la proteína Spike. En los últimos meses, C.4 ha acumulado nuevas mutaciones conforme se ha transmitido en Lima, Callao y Amazonas.
8/13
8/13
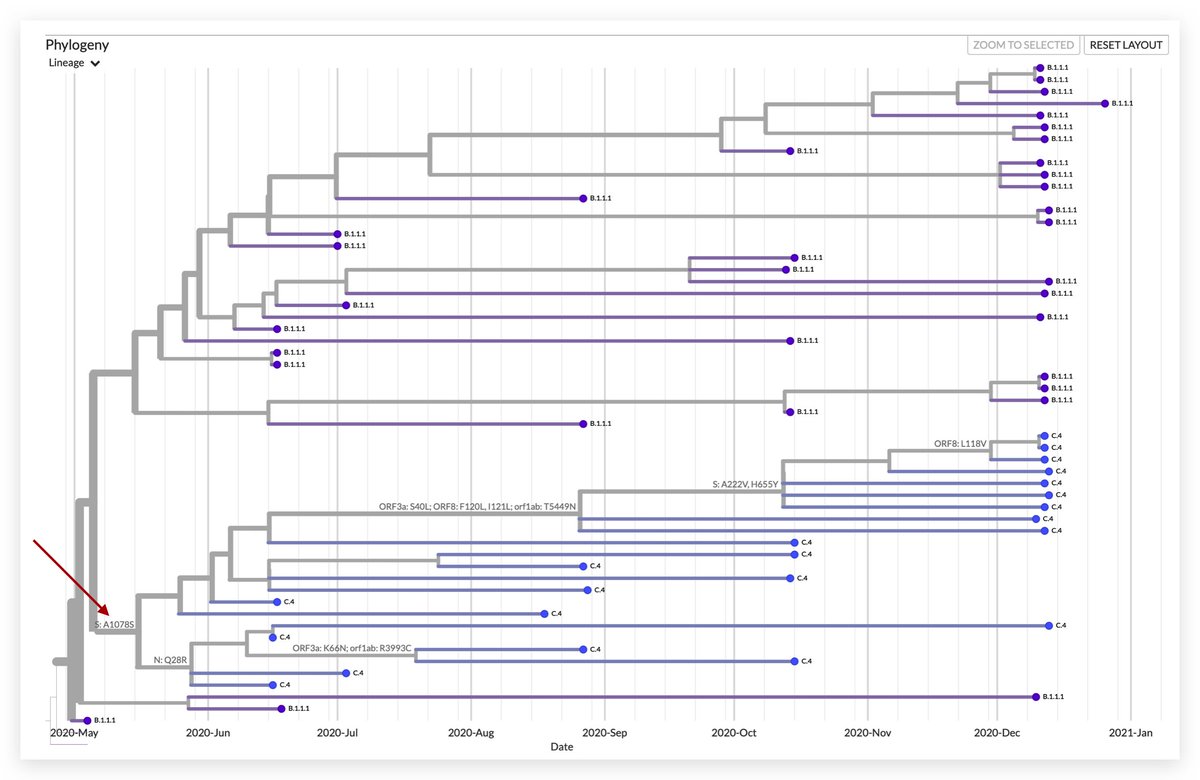
En diciembre observamos que 7 genomas C.4 habían desarrollado dos nuevas mutaciones en la proteína Spike. La primera, A222V, es la misma que observamos en la variante europea B.1.177 🇪🇸.
9/13
9/13

Una mutación que aparece independientemente en distintas partes del mundo indica que *podría* conferirle una ventaja adaptativa sobre el resto de variantes. A este fenómeno le llamamos 'evolución convergente'.
10/13
10/13
https://twitter.com/quipupe/status/1352346098950680579
La 2da mutación, H655Y, suena en los últimos días porque está en la variante P.1 🇧🇷 Los genomas peruanos NO son iguales a los de Manaos, sino que la mutación ha aparecido de forma independiente en C.4 🇵🇪 y P.1 🇧🇷. Segundo caso de evolución convergente en una misma variante.
11/13
11/13
Por ahora no hay evidencia que B.1.117 o C.4 sean más transmisible o letales que el resto de variantes que tenemos circulando. Necesitamos más estudios epidemiológicos y de laboratorio para confirmar esa hipótesis.
12/13
12/13
Vuelvo a insistir: debemos fortalecer la vigilancia genómica en Perú para detectar las nuevas variantes locales e importadas que aparezcan en los próximos meses. El virus seguirá cambiando y debemos seguirle el rastro. #SinCienciaNoHayFuturo
13/13
larepublica.pe/ciencia/2021/0…
13/13
larepublica.pe/ciencia/2021/0…
PS. Sigan a @quipupe para más info sobre las variantes peruanas.
https://twitter.com/quipupe/status/1350555016726196226?s=20
• • •
Missing some Tweet in this thread? You can try to
force a refresh








