Concytec financiará el proyecto de #VigilanciaGenomica de SARS-CoV-2. ¡Gracias! Los fondos permitirán mantener el equipo de trabajo y secuenciar 1,500 genomas en 2021 en laboratorios académicos de Lima, Arequipa y Chachapoyas. 🧵
1/12
bit.ly/3ct1KgE
1/12
bit.ly/3ct1KgE

2/12
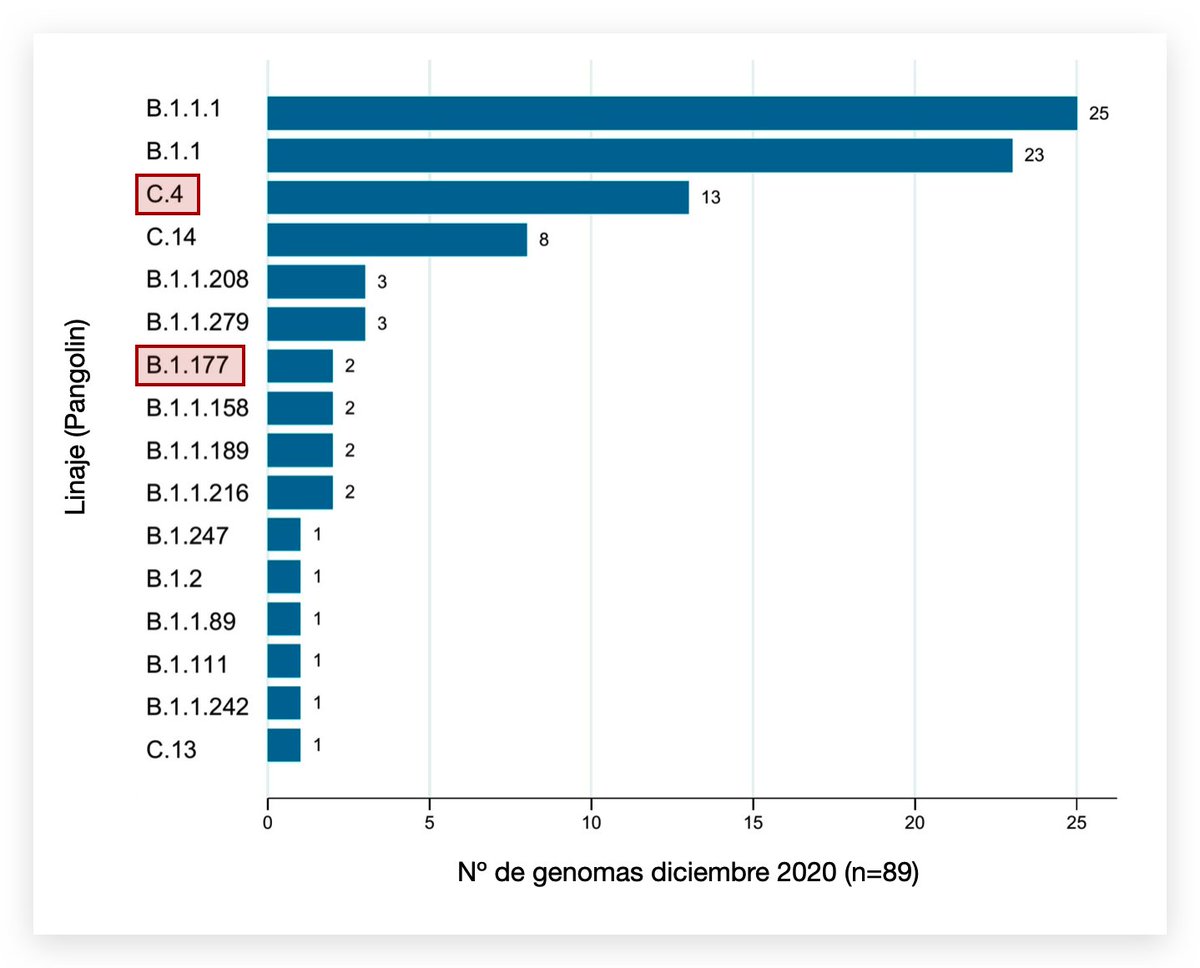
Entre marzo 2020 y enero 2021, INS y UPCH han generado 970+ genomas de casos en 16 regiones. Reportamos 49 linajes circulantes, incluyendo B.1.1.7 🇬🇧 y P.1 🇧🇷. Pueden explorar los datos en la página de @nextstrain que mantiene @quipupe
bit.ly/3bKfe8w
Entre marzo 2020 y enero 2021, INS y UPCH han generado 970+ genomas de casos en 16 regiones. Reportamos 49 linajes circulantes, incluyendo B.1.1.7 🇬🇧 y P.1 🇧🇷. Pueden explorar los datos en la página de @nextstrain que mantiene @quipupe
bit.ly/3bKfe8w
3/12
B.1.1.7 🇬🇧 se ha reportado en 98 países. Es 1.5x más transmisible y parece estar asociado a mayor severidad + riesgo de muerte. Tenemos 3 casos reportados en Lima en enero.
nyti.ms/3voSdjf

B.1.1.7 🇬🇧 se ha reportado en 98 países. Es 1.5x más transmisible y parece estar asociado a mayor severidad + riesgo de muerte. Tenemos 3 casos reportados en Lima en enero.
nyti.ms/3voSdjf


4/12
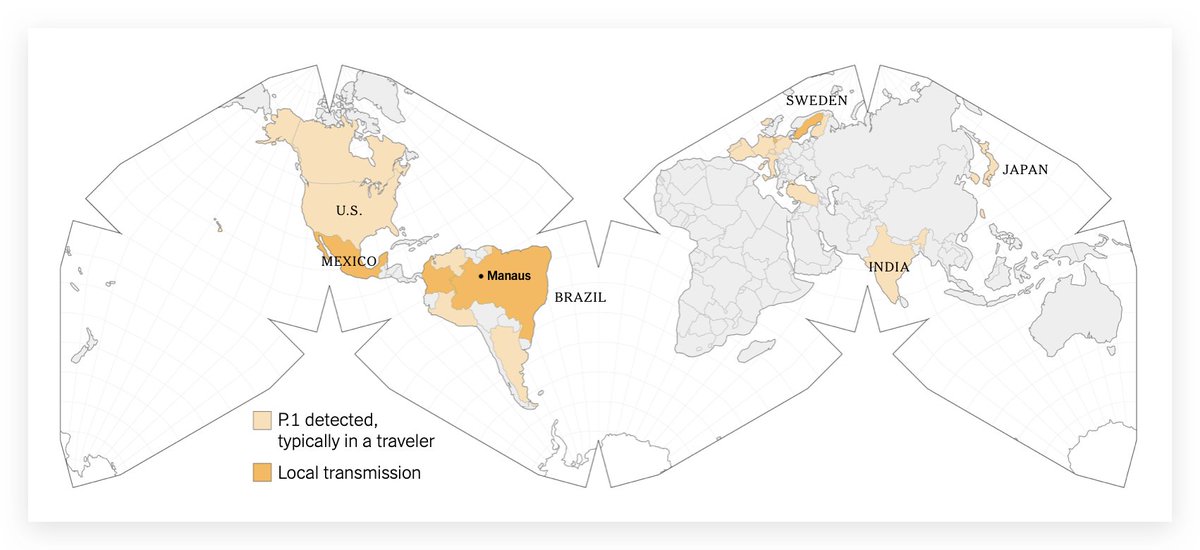
P.1 🇧🇷 se reporta en 24 países. Estudios preliminares indican que es 2x más transmisible y puede escapar (parcialmente) la neutralización por anticuerpos. Esto le haría capaz de reinfectar a personas previamente inmunizadas. 23 casos reportados en Lima, Huánuco y Loreto.

P.1 🇧🇷 se reporta en 24 países. Estudios preliminares indican que es 2x más transmisible y puede escapar (parcialmente) la neutralización por anticuerpos. Esto le haría capaz de reinfectar a personas previamente inmunizadas. 23 casos reportados en Lima, Huánuco y Loreto.

5/12
Es probable que B.1.1.7 y P.1 ya se transmitan activamente en varias regiones del país. La reciente relajación de medidas para viajes nacionales e internacionales llevaría a más introducciones y transmisión en las próximas semanas. @ocram
Es probable que B.1.1.7 y P.1 ya se transmitan activamente en varias regiones del país. La reciente relajación de medidas para viajes nacionales e internacionales llevaría a más introducciones y transmisión en las próximas semanas. @ocram
6/12
Pero no basta saber que ya están aquí. Necesitamos saber en qué regiones ya tenemos transmisión comunitaria. Al ser más transmisibles, se espera que en los próximos meses aumenten su frecuencia y empiecen a dominar sobre variantes locales.
Pero no basta saber que ya están aquí. Necesitamos saber en qué regiones ya tenemos transmisión comunitaria. Al ser más transmisibles, se espera que en los próximos meses aumenten su frecuencia y empiecen a dominar sobre variantes locales.
7/12
Esto pasó en Dinamarca (que secuencia 3,000+ genomas por semana): se reportó la primera introducción de B.1.1.7 🇬🇧 a mediados de diciembre y hoy es casi el 85% de los casos en el país.

Esto pasó en Dinamarca (que secuencia 3,000+ genomas por semana): se reportó la primera introducción de B.1.1.7 🇬🇧 a mediados de diciembre y hoy es casi el 85% de los casos en el país.
https://twitter.com/MadsAlbertsen85/status/1370002101657612291

8/12
Por eso debemos realizar muestreos frecuentes y aleatorios en todas regiones para monitorear la transmisión y evolución de estas variantes en Perú.
Por eso debemos realizar muestreos frecuentes y aleatorios en todas regiones para monitorear la transmisión y evolución de estas variantes en Perú.
9/12
Urge también caracterizar variantes locales que ya circulan por aquí. Y también a nuevas variantes que puedan aparecer en los próximos meses en respuesta a las vacunas y el gran número de individuos infectados.
Urge también caracterizar variantes locales que ya circulan por aquí. Y también a nuevas variantes que puedan aparecer en los próximos meses en respuesta a las vacunas y el gran número de individuos infectados.
https://twitter.com/pablotsukayama/status/1352468136742809603
10/12
La información sobre variantes circulantes debería ser parte de los indicadores usados por el Gobierno para establecer el nivel de alerta de cada región.

La información sobre variantes circulantes debería ser parte de los indicadores usados por el Gobierno para establecer el nivel de alerta de cada región.


11/12
Si bien este financiamiento es un avance, 1,500 genomas en un año es una fracción muy pequeña del total de infecciones en 2021. Desde enero reportamos más de 1,500 nuevos casos (PCR+) *por día*. Deberíamos secuenciar más, y para eso se necesita mayor inversión.
Si bien este financiamiento es un avance, 1,500 genomas en un año es una fracción muy pequeña del total de infecciones en 2021. Desde enero reportamos más de 1,500 nuevos casos (PCR+) *por día*. Deberíamos secuenciar más, y para eso se necesita mayor inversión.
12/12
Llevamos un año de pandemia y solo Concytec ha apostado por la vigilancia genómica. Dice mucho sobre la confianza del país en soluciones basadas en ciencia. Pero vamos con todo y seguiremos informando. Gracias por su apoyo.
#SinCienciaNoHayFuturo ✊
Llevamos un año de pandemia y solo Concytec ha apostado por la vigilancia genómica. Dice mucho sobre la confianza del país en soluciones basadas en ciencia. Pero vamos con todo y seguiremos informando. Gracias por su apoyo.
#SinCienciaNoHayFuturo ✊
13/n
¡Gracias por su apoyo! Aprovecho su atención para comentarles sobre los intentos (fallidos) de financiar este proyecto a través de fondos privados. Tal vez nos puedan ayudar a mejorar nuestra estrategia o a encontrar donantes interesadxs.
¡Gracias por su apoyo! Aprovecho su atención para comentarles sobre los intentos (fallidos) de financiar este proyecto a través de fondos privados. Tal vez nos puedan ayudar a mejorar nuestra estrategia o a encontrar donantes interesadxs.
https://twitter.com/pablotsukayama/status/1366587106986975234
14/n
¿Por qué necesitamos más fondos? Los casi 1,000 genomas generados en 2020 son menos del 0.1% de casos oficiales. En UK procesan un 8%. En Australia, casi 60%. Debemos apuntar al menos a un 1% para asegurar un rastreo adecuado del virus en 2021.
¿Por qué necesitamos más fondos? Los casi 1,000 genomas generados en 2020 son menos del 0.1% de casos oficiales. En UK procesan un 8%. En Australia, casi 60%. Debemos apuntar al menos a un 1% para asegurar un rastreo adecuado del virus en 2021.
15/n
1% de casos = 300-800 genomas por mes. El financiamiento de Concytec (S/800,000) nos da para unos 120-150 por mes. Necesitamos al menos S/2 millones para llegar a ese 1%.
1% de casos = 300-800 genomas por mes. El financiamiento de Concytec (S/800,000) nos da para unos 120-150 por mes. Necesitamos al menos S/2 millones para llegar a ese 1%.
16/n
Hace meses que insistimos que el Gobierno debe financiar estas actividades. La vigilancia genómica ya dejó de ser un proyecto de investigación y se ha convertido en una herramienta de salud pública que nos ayuda a entender mejor la transmisión y evolución local del virus.
Hace meses que insistimos que el Gobierno debe financiar estas actividades. La vigilancia genómica ya dejó de ser un proyecto de investigación y se ha convertido en una herramienta de salud pública que nos ayuda a entender mejor la transmisión y evolución local del virus.
17/n
MINSA debe fortalecer y financiar al INS para que aumente su capacidad de vigilancia. Pero no deben ser los únicos. Desde las universidades podemos complementar estos esfuerzos de forma flexible, eficiente y descentralizada.
MINSA debe fortalecer y financiar al INS para que aumente su capacidad de vigilancia. Pero no deben ser los únicos. Desde las universidades podemos complementar estos esfuerzos de forma flexible, eficiente y descentralizada.
18/n
Hace un año sufrimos por falta de laboratorios de biología molecular para hacer pruebas de PCR. Las universidades ofrecieron sus laboratorios y no se les tomó cuenta. No repitamos esa historia.
Hace un año sufrimos por falta de laboratorios de biología molecular para hacer pruebas de PCR. Las universidades ofrecieron sus laboratorios y no se les tomó cuenta. No repitamos esa historia.
19/n
Desde hace meses converso con fundaciones, sociedades, fondos de inversión, farmacéuticas, agroexportadoras, mineras, bancos y la misma @confiep. Todos te dan una palmada en el hombro, te felicitan por el esfuerzo y te dan una de tres respuestas:
Desde hace meses converso con fundaciones, sociedades, fondos de inversión, farmacéuticas, agroexportadoras, mineras, bancos y la misma @confiep. Todos te dan una palmada en el hombro, te felicitan por el esfuerzo y te dan una de tres respuestas:
20/n
(1) No nos llames. Nosotros te llamamos.
(2) La empresa ha tenido muchas pérdidas en 2020 y no puede apoyar en este momento.
(3) Ya hemos donado a otras iniciativas y no podemos aportar más. Sorry.
(1) No nos llames. Nosotros te llamamos.
(2) La empresa ha tenido muchas pérdidas en 2020 y no puede apoyar en este momento.
(3) Ya hemos donado a otras iniciativas y no podemos aportar más. Sorry.
21/n
A diferencia de otras iniciativas que han recibido apoyo privado (EPP, oxígeno, respiradores, pruebas moleculares y vacunas made in Peru), la vigilancia genómica no genera un producto tangible o comercializable.
A diferencia de otras iniciativas que han recibido apoyo privado (EPP, oxígeno, respiradores, pruebas moleculares y vacunas made in Peru), la vigilancia genómica no genera un producto tangible o comercializable.
22/n
Generamos información epidemiológica basada en datos abiertos. Es difícil vender esta idea a un donante privado que busca un retorno a su inversión.
Generamos información epidemiológica basada en datos abiertos. Es difícil vender esta idea a un donante privado que busca un retorno a su inversión.
23/n
Por supuesto, estas empresas no tienen por qué donar si no les resulta conveniente. Pero yo pensaba que las grandes empresas ayudaban a resolver los grandes problemas del país. Al menos eso me dijeron en el colegio.
Por supuesto, estas empresas no tienen por qué donar si no les resulta conveniente. Pero yo pensaba que las grandes empresas ayudaban a resolver los grandes problemas del país. Al menos eso me dijeron en el colegio.
24/n
¿Cómo hacer? ¿Ideas?
¿Cómo hacer? ¿Ideas?
• • •
Missing some Tweet in this thread? You can try to
force a refresh