Esta semana reportamos la identificación de un nuevo linaje (variante) de SARS-CoV-2 que parece expandirse rápidamente en Perú y Chile. Le llamamos C.37. Les cuento lo que sabemos y no sabemos al respecto. 🧵
1/25
1/25

El anuncio fue publicado en virological.org, donde virólogos de todo el mundo discuten sus resultados preliminares. No es un pre-print, no ha sido revisado por pares y requerimos más datos para verificar estas observaciones. bit.ly/3gyZZBZ
2/
2/
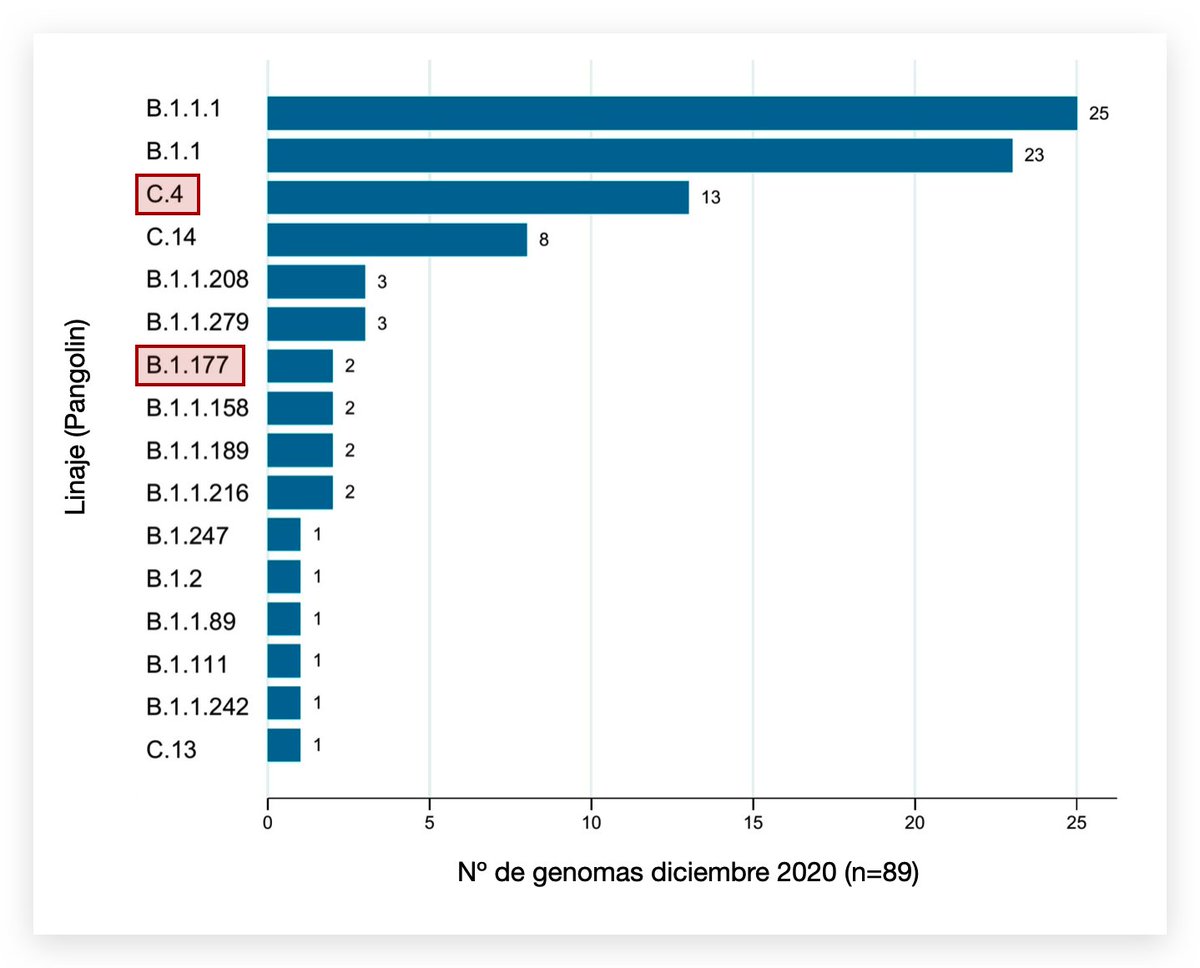
C.37 desciende de un linaje llamado B.1.1.1 que circula por todo el mundo desde inicios de la pandemia. En Perú, identificamos a B.1.1.1 en 10.2% de los 1,030 genomas procesdos en 2020. B.1.1.1 no es la variante B.1.1.7 🇬🇧 y tiene un origen diferente a P.1 🇧🇷
3/
3/

A fines de diciembre vemos en Lima una variante descendiente de B.1.1.1 que acumula varias mutaciones nuevas en su genoma, incluyendo algunas que ya hemos visto en las variantes de preocupación (VOCs) B.1.1.7 🇬🇧, B.1.351 🇿🇦, P.1 🇧🇷 #convergenciaevolutiva
4/
4/
En 2021, esta nueva variante (C.37) se expande rápidamente y ahora la vemos en 50 de 123 (40.6%) genomas reportados en Lima y Callao entre enero y marzo. En ese mismo periodo, en Lima y Callao, solo observamos 3 genomas P.1🇧🇷 (2.9%)
5/
5/
Qué la diferencia de las 50+ variantes que ya tenemos en Perú? 1) Tiene una deleción que ya hemos visto en las variantes de preocupación 🇬🇧🇧🇷🇿🇦 2) Presenta mutaciones que podrían ayudarle a evadir la neutralización por anticuerpos; 3) Preocupa su rápido crecimiento en Lima.
6/
6/

¿Pero, no que la variante P.1 🇧🇷 ya dominaba en todo Lima? En febrero-marzo, INS analizó 199 muestras de Lima e identificó la variante P.1 en 79 (39.7%). Lejos del 2.9% que nosotros reportamos pero cerca del 40.6% que tenemos para C.37. Algo no cuadra.
7/ web.ins.gob.pe/index.php/es/p…
7/ web.ins.gob.pe/index.php/es/p…
Para este análisis, INS no utilizó el secuenciamiento de genoma completo, sino una alternativa de menor costo, basada en PCR, que permite la detección de las 3 variantes de preocupación en cientos de muestras a la vez.
8/
medrxiv.org/content/10.110…
8/
medrxiv.org/content/10.110…
Sin embargo, este ensayo no es tan preciso como el secuenciamiento de genoma completo, ya que solo identifica mutaciones en pequeñas regiones del virus. Estas mutaciones podrían estar presentes en otras variantes que no conocemos aún. #convergenciaevolutiva
9/
9/
C.37 y P.1 comparten la mutación ORF1a:3675-3677 y (en principio) darían el mismo resultado en esta prueba de PCR. O sea: es posible que muchas de las muestras identificadas como P.1 por INS sean realmente C.37.
10/
10/
Este mismo ensayo se aplicó a 380 muestras adicionales de otras 9 regiones en marzo-abril. Reportaron que 1/3 de ellas corresponden a P.1. Explican que habría ingresado multiples veces por la frontera con Brasil, llega hasta Lima y luego se disemina a otras regiones.
11/
11/

O será que una variante local (C.37) se genera en Lima, se expande y luego se transmite a otras regiones? No los sabemos aún. La única manera de confirmar es secuenciando un grupo de muestras ya identificadas como P.1 por PCR. Ya estamos coordinando ese experimento con INS.
12/
12/
¿Tienen más evidencia para apoyar este argumento? ¡Tenemos! A fines de enero empezamos a observar casos de C.37 en Chile y EEUU. En abril ya se reportan 160 en Chile y 133 en EEUU. Hoy la vemos en Argentina, Brasil, Ecuador, México, Alemania, España, Francia, UK y Australia.
13/
13/
Con los datos disponibles, no podemos asegurar si C.37 se originó en Perú o en Chile. Ambos tienen epidemias similares en 2021 y comparten muchos vuelos diarios. Otra posibilidad es que se haya introducido a Chile y Perú desde otro país de la región que aún no la detecta.
14/

14/


Nuevamente: Esta información es preliminar y nuestras ideas sobre su origen y el peligro que representa pueden cambiar en las próximas semanas con nueva información. Mantendremos la vigilancia para seguirle el rastro a C.37, P.1, B.1.1.7 y otras variantes que aparezcan.
15/
15/
¿Es C.37 más transmisible o más virulenta que otras variantes? No lo sabemos aún. Necesitamos colectar más datos y diseñar estudios que nos permitan responder esas preguntas. Esto va requerir tiempo, fondos y nuevas colaboraciones locales e internacionales.
16/
16/
¿Es responsable del aumento de casos y muertes en la 2da ola? No lo sabemos. Pero la aparición y expansión de este linaje parecen cuadrar con el aumento de casos en varias regiones. Pero recuerden que correlación no implica causalidad.
17/
17/

¿Las vacunas serán eficaces ante esta variante? No lo sabemos. Esto requiere de muchos experimentos y análisis adicionales. Pero C.37 presenta mutaciones en regiones asociadas (en el lab) a escape de neutralización por anticuerpos.
18/
doi.org/10.1016/j.cell…
18/
doi.org/10.1016/j.cell…
¿Es peor tener P.1 o C.37? Ambas opciones parecen ser muy malas para el país. Igual que nuestra 2da vuelta electoral 🙃 🇵🇪. De P.1 sabemos que es más transmisible y que podría tener mayor capacidad de reinfección. De C.37 no sabemos nada aún.
19/
19/
Entonces, para qué sirve esta información? 1) Desde un punto de vista epidemiológico, es importante entender cómo se inició esta expansión y qué propiedades tienen las epidemias asociadas a distintas variantes.
20/
20/
2) Porque nos permite alertar a otros países y a la comunidad científica internacional. 3) Porque ayuda a informar el diseño de nuevas vacunas y tratamientos.
21/
21/
En resumen: Hay mucho que no sabemos aún sobre C.37. Pero lo que sabemos, preocupa. Y debemos corroborar las cifras que INS ha reportado en las últimas semanas.
22/
22/
Este estudio es posible gracias al financiamiento de @ConcytecPeru y a los datos compartidos *abiertamente* por varias instituciones y países en @GISAID. Gracias a ellos podremos seguir el rastro de esta variante en los próximos meses.
23/
nature.com/articles/d4158…
23/
nature.com/articles/d4158…
Reitero: Es necesario fortalecer la vigilancia genómica en Perú. Pasa por fortalecer al INS pero también por invertir en descentralizar estas investigaciones, fortalecer universidades y promover el desarrollo de científicos en todo el país.
24/
24/
https://twitter.com/pablotsukayama/status/1383478058129129480
A quienes nos gobiernan y lxs que lo harán desde julio: Por favor, escuchen a sus científicos y tomen decisiones sobre la pandemia basados en ciencia + evidencia. Queremos y podemos ayudar. #SinCienciaNoHayFuturo
25/
25/
• • •
Missing some Tweet in this thread? You can try to
force a refresh