
Le preprint de notre analyse des #Ct des tests #RTPCR de #SARSCoV2, en partenariat avec la Société Française de Microbiologie et plus de 20 laboratoires de virologie français est maintenant en ligne. **NON RELU PAR LES PAIRS**
medrxiv.org/content/10.110…
1/N
medrxiv.org/content/10.110…
1/N
Nous y analysons des valeurs quantitatives de millions de tests #RTPCR effectués en France en 2020 : les Ct, ou cycles de doublement.
assets.publishing.service.gov.uk/government/upl… (doc très clair en anglais)
publichealthontario.ca/-/media/docume… (doc en français)
**NON RELU PAR LES PAIRS**
2/N
assets.publishing.service.gov.uk/government/upl… (doc très clair en anglais)
publichealthontario.ca/-/media/docume… (doc en français)
**NON RELU PAR LES PAIRS**
2/N

Il y a eu un débat sur ces Ct et l'opportunité de les communiquer aux patients.
Primo, cela dépend de l'échantillon. Secundo, pour les #coronavirus, il est risqué de voir dans le nombre de copies d'ARN une charge virale.
osf.io/5gra3/
**NON RELU PAR LES PAIRS**
3/N
Primo, cela dépend de l'échantillon. Secundo, pour les #coronavirus, il est risqué de voir dans le nombre de copies d'ARN une charge virale.
osf.io/5gra3/
**NON RELU PAR LES PAIRS**
3/N

Nos travaux portent sur des tests #RTPCR sur #SARSCOV2 réalisés chez environ 2 millions de personnes dans toute la France. On voit une hétérogénéité spatiale mais aussi temporelle (qui suit la dynamique de l'épidémie).
**NON RELU PAR LES PAIRS**
4/N
**NON RELU PAR LES PAIRS**
4/N

Nous confirmons que les valeurs de #Ct sont très variables. Par exemple, il y a un effet laboratoire mais aussi un effet du kit #RTPCR utilisé.
On voit que sur 12 kits comparés les gènes ciblés semblent aussi importer.
**NON RELU PAR LES PAIRS**
5/N
On voit que sur 12 kits comparés les gènes ciblés semblent aussi importer.
**NON RELU PAR LES PAIRS**
5/N
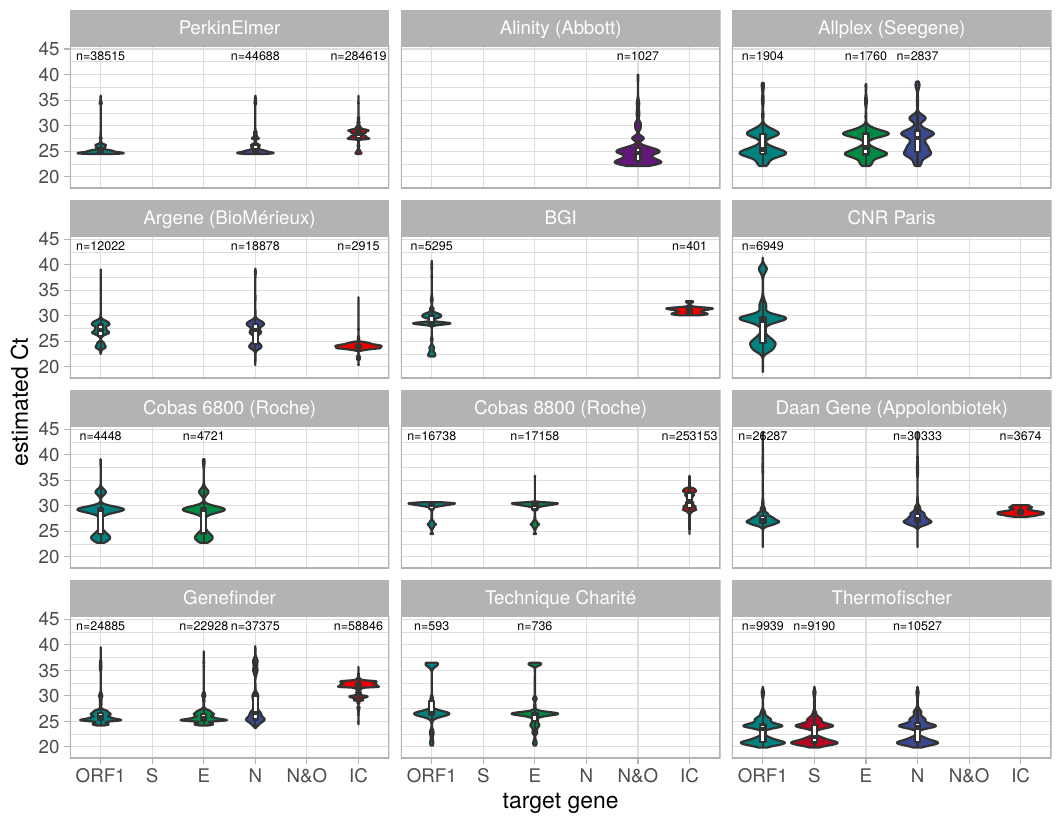
Cet effet "cible" est significatif et assez joli (pour un virologue). Que les #Ct de cibles #RTPCR dans le gène N soient plus faibles que ceux dans l'#ORF1 est cohérent avec le fait que pour les #coronavirus il existe plus de copies du coté 3'.
**NON RELU PAR LES PAIRS**
6/N
**NON RELU PAR LES PAIRS**
6/N

On trouve aussi que plus il s'est écoulé de jours depuis le début des symptômes, plus les #Ct sont élevés, ce qui est cohérent avec la cinétique de l'infection (cf. par exemple les travaux de l'équipe de Jérémie Guedj).
pnas.org/content/118/8/…
**NON RELU PAR LES PAIRS**
7/N
pnas.org/content/118/8/…
**NON RELU PAR LES PAIRS**
7/N

On détecte une baisse des #Ct avec l'âge. En revanche, l'effet du sexe n'est pas significatif.
Ce résultat est intéressant car la variation en fonction de l'âge est débattue. Mais la plupart des analyses existantes n'étaient pas multi-variées.
**NON RELU PAR LES PAIRS**
8/N
Ce résultat est intéressant car la variation en fonction de l'âge est débattue. Mais la plupart des analyses existantes n'étaient pas multi-variées.
**NON RELU PAR LES PAIRS**
8/N

Mais l'effet frappant est le lien entre #Ct et nombre de reproduction #Rt de l'épidémie au moment du prélèvement.
Cet effet est prédit par la théorie (si l'épidémie décroît, on détecte des infections plus anciennes).
**NON RELU PAR LES PAIRS**
9/N
Cet effet est prédit par la théorie (si l'épidémie décroît, on détecte des infections plus anciennes).
**NON RELU PAR LES PAIRS**
9/N

Nous avons testé si l'utilisation des valeurs de #Ct améliore la prévision du #Rt à l'échelle d'une semaine avec des techniques #ARIMA.
La réponse est oui, surtout aux moments où il y a de grandes variations.
**NON RELU PAR LES PAIRS**
10/N
La réponse est oui, surtout aux moments où il y a de grandes variations.
**NON RELU PAR LES PAIRS**
10/N

En résumé, les valeurs de #Ct sont très variables et, à un niveau individuel, peu interprétables.
Mais, au niveau de la population, on confirme des effets biologiques connus ou débattus, et on montre que ces Ct sont utiles en santé publique.
**NON RELU PAR LES PAIRS**
11/N
Mais, au niveau de la population, on confirme des effets biologiques connus ou débattus, et on montre que ces Ct sont utiles en santé publique.
**NON RELU PAR LES PAIRS**
11/N
• • •
Missing some Tweet in this thread? You can try to
force a refresh









